Onkologische Forschungslabore
Im Comprehensive Cancer Center Ostbayern (CCCO) befassen sich verschiedene Arbeitsgruppen und Labore aus Medizinern und Wissenschaftlern mit Fragestellungen zur Diagnose und Behandlung im onkologischen Bereich.
Unsere onkologischen Forschungslabore
AG Algorithmen und Supercomputing in der Onkologie
Leitung der Projekte:
- Prof. Dr. Andreas Schäfer
- Prof. Dr. Tilo Wettig
Mitglieder der Arbeitsgruppe:
- Dr. Jacques Bloch
- Peter Georg
- Dr. Robert Lohmayer
- Dr. Nils Meyer
- Emmanuel Nowak
- Daniel Richtmann
- Julia Roigk
- Dr. Stefan Solbrig
Tumorgenome spiegeln die Geschichte einer Krebserkrankung wieder, erlauben aber auch Vorhersagen für die weitere Entwicklung der Krankheit. Sie können damit nützlich für Therapieentscheidungen sein. Genomische Daten sind hoch komplex, und um sie optimal zu nutzen, brauchen wir immer effizientere Algorithmen, Implementierungen und spezialisierte Architekturen für Hochleistungsrechner. Unsere Arbeitsgruppe kommt aus der Theoretischen Elementarteilchenphysik und hat langjährige Expertise im massiv parallelen Höchstleistungsrechnen (Supercomputing), inklusive der Entwicklung applikationsspezifischer Hard- und Software in Zusammenarbeit mit namhaften Firmen (z.B. IBM und Intel). Seit 2016 arbeitet die Arbeitsgruppe auch auf dem Gebiet der Onkologie, in Kooperation mit den Arbeitsgruppen von Rainer Spang, Philipp Beckhove (RCI) und Christoph Klein (ITEM-R). Der Schwerpunkt liegt dabei auf der massiven Beschleunigung von Machine Learning Algorithmen (z.B. für Krebsprogressionsmodelle), auf der Entwicklung neuer bzw. verbesserter Algorithmen (z.B. für die Analyse von Einzelzellsequenzierungen) und auf dem Einsatz spezieller Hardware-Beschleuniger (z.B. FPGAs). Dadurch können existierende Algorithmen um mehrere Größenordnungen beschleunigt werden und neue bioinformatische Probleme in Angriff genommen werden, die mit bisherigen Methoden nicht zugänglich waren.
Etablierte Methoden
- High-Performance Computing
- Algorithmen-Entwicklung
- Co-Design von applikationsspezifischer Hard- und Software
- FPGA-Programmierung
Methoden in Entwicklung
- Tensorapproximationen für Krebsprogressionsmodelle
- Fehlerkorrektur-Algorithmen für Einzelzellsequenzierung
- Parallele Implementierung von Machine Learning Algorithmen
Laufende Forschungsprojekte
- SFB/TRR-55
- KONWIHR-III: Simulationssoftware für Krebsprogressionsmodelle auf Multi-/Manycore-Systemen, zusammen mit Rainer Spang und Niko Beerenwinkel (ETH Zürich)
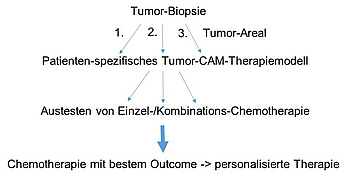
AG Aung/Härteis - Translationale Sarkomforschung: Patientenspezifisches Tumor-CAM-Therapiemodell
Leitung der Arbeitsgruppe:
- Prof. Dr. med. Thiha Aung, Lehrstuhl für Molekulare und Zelluläre Anatomie, Universität Regensburg
- Prof. Dr. Silke Härteis, Professur für Molekulare und Zelluläre Anatomie, Universität Regensburg
Mitglieder der Arbeitsgruppe:
- Prof. Dr. Ralph Witzgall, Lehrstuhl für Molekulare und Zelluläre Anatomie, Universität Regensburg
- Prof. Dr. Dr. Lukas Prantl, Leiter der Abteilung für Plastische-, Hand- und Wiederherstellungschirurgie, Universitätsklinikum Regensburg
- Lucia Denk, Medizinisch-Technische Assistentin
Prof. Dr. Thiha Aung hat langjährige Erfahrung im Einsatz des konventionellen (in ovo) bzw. später von ihm modifizierten (ex ovo) Chorion-Allantois-Membran (CAM)-Modells. Um die Effekte von Chemotherapie, Strahlentherapie, Tumorstammzellen und deren Kommunikation in der Tumornische in vivo zu untersuchen, hat Herr Prof. Dr. Aung ein CAM-Modell etabliert und erfolgreich bei verschiedenen Tumormodellen (Diffuse large B-cell lymphoma, Glioblastome, Plattenepithelkarzinome) eingesetzt (Koch et al., 2016; Koch et al., 2014; Sperling et al., 2017). Beim in ovo CAM-Modell steht zur Gabe des Chemotherapeutikums bzw. der Strahlentherapie jedoch nur eine ca. 1 x 1 cm große Öffnungsstelle der Eierschale zur Verfügung. Dadurch wird die Gabe von Chemotherapie stark behindert und eine gezielte Strahlentherapie kann nur unter erschwerten Bedingungen durchgeführt werden. Aufgrund dieser Problematik hat Herr Prof. Dr. Aung das in ovo CAM-Modell modifiziert und ein ex ovo CAM-Modell entwickelt (Kauffmann et al., 2018; Kauffmann et al., 2017). Die Optimierung des CAM-Modells umfasst einen größeren Arbeitsbereich auf der Chorion-Allantois-Membran. Außerdem können in diesem optimierten Modell die verschiedenen Areale eines Tumors gleichzeitig analysiert werden sowie die Ansprechrate von Chemotherapie, Strahlentherapie und Immuntherapie beobachten werden.
Frau Prof. Dr. Silke Härteis hat langjährige Erfahrung in der Durchführung von Mikroinjektionen bei Xenopus laevis Oozyten. Diese Methode ist entscheidend bei der Applikation der Chemotherapeutika im modifizierten CAM-Assay. Die häufigsten Resistenzmechanismen bei Chemotherapie entwickeln sich über die Expression von ABC-/MDR-Transportern, die im Oozytenexpressionssystem exprimiert und untersucht werden können. Das CAM-Modell wurde in der Arbeitsgruppe von Frau Prof. Härteis am Lehrstuhl für Molekulare und Zelluläre Anatomie etabliert. Der Lehrstuhl bietet eine hervorragende Infrastruktur und methodische Expertise für die histologische Aufarbeitung und die bildgebenden Verfahren.
Etablierte Methoden
Modifiziertes ex ovo Chorion-Allantois-Membran (CAM)-Modell
Nacktmäuse dienen seit Jahrzenten als Tiermodell zur Untersuchung der Tumorbiologie. Bislang ist es nicht gelungen, die Zahl der hierfür eingesetzten Tiere substantiell zu reduzieren. Wir beabsichtigen, exemplarisch am Modell der Weichteilsarkome, das modifizierte ex ovo CAM-Modell als leistungsstarke und effiziente Alternativmethode zu etablieren. Dadurch sollte es gelingen, die Zahl der bislang in diesem Gebiet verwendeten Nacktmäuse deutlich zu verringern.
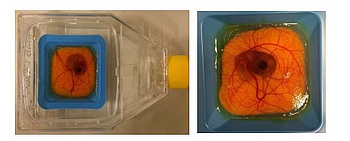
Zur Vorbereitung des CAM (Chorion-Allantois-Membran)-Assays werden befruchtete Eier in einem Brutschrank (Luftfeuchtigkeit 65 %, Temperatur 37°C) für 4 Tage bei stündlicher Rotation inkubiert. Am 4. Tag wird die Eihülle geöffnet. Die Schale wird mit einer scharfen Pinzette vorsichtig angehoben und die obere Membran wird langsam abgehoben, so dass die Chorion-Allantois-Membran sichtbar wird. Nach weiteren 4 Tagen werden die unterschiedlichen Sarkom-Zelllinien auf die Chorion-Allantois-Membran aufgetragen. Anschließend wird das befruchtete Ei in eine Zellkulturflasche überführt und mit sterilem Wasser umgeben (s. Abb. 1). Alle 24 Stunden wird das Wachstum der Tumoren mittels 3D-Mikroskopie analysiert. Am 16. Tag werden die Tumore aus dem CAM-Assay entnommen, gewogen, in Paraffin eingebettet und histologisch untersucht.
Das Ziel der Arbeitsgruppe ist es, die Effekte von Chemotherapie, Strahlentherapie und Immuntherapie im modifizierten ex ovo CAM-Modell auf die Sarkome zu übertragen. Folgende Aspekte sollen im Einzelnen bearbeitet werden: Untersuchung von unterschiedlichen Tumorarealen (Tumordiversitäten) aus Primärmaterial in Bezug auf
- Proliferation
- Infiltration
- Angiogenese (Pion et al., Microvascular Research, 2021)
- Ansprechverhalten auf Chemotherapie
- Metastasierung
Zusammenfassend sollen die Untersuchungen unter Verwendung eines Patienten-spezifischen Tumor-CAM-Therapiemodells zu einem besseren Verständnis der Tumorbiologie beitragen, um eine personalisierte medikamentöse Therapie zu entwickeln (s. Abb. 2).
Laufende Forschungsprojekte
- Modifikation des Chorion-Allantois-Membran (CAM)-Modells für ein besseres Verständnis der Tumordiversitäten und zur Austestung neuer medikamentöser Therapien bei Osteosarkomen – Patienten-spezifisches Tumor-CAM-Therapiemodell.
- Modifikation des Chorion-Allantois-Membran (CAM)-Modells für ein besseres Verständnis der Tumordiversitäten und zur Austestung neuer medikamentöser Therapien bei Kindersarkomen (Ewing-Sarkom; Rhabdomyosarkom) – Patienten-spezifisches Tumor-CAM-Therapiemodell.
-
Modifikation des Chorion-Allantois-Membran (CAM)-Modells für ein besseres Verständnis der Tumordiversitäten und zur Austestung neuer medikamentöser Therapien bei Angiosarkomen – Patienten-spezifisches Tumor-CAM-Therapiemodell.
Kooperationen
- Prof. Dr. Wolfgang Herr, Klinik und Poliklinik für Innere Medizin III und Leiter des Sarkomzentrums, Universitätsklinikum Regensburg
- Prof. Dr. Hinrich Abken, RCI Lehrstuhl für Gen-Immuntherapie, Universität Regensburg
- Prof. Dr. Mark Berneburg, Direktor der Klinik und Poliklinik für Dermatologie, Universitätsklinikum Regensburg
- Prof. Dr. Selim Corbacioglu, Abteilung für Pädiatrische Hämatologie, Onkologie und Stammzelltransplantation, Universitätsklinikum Regensburg
- Dr. Katja Evert / Prof. Dr. Matthias Evert, Institut für Pathologie, Universität Regensburg
- Dr. David Braig / Prof. Dr. Steffen Eisenhardt, Klinik für Plastische und Handchirurgie, Universitätsklinikum Freiburg
- Dr. Carolin Knebel / Prof. Dr. Rüdiger von Eisenhart-Rothe, Klinik und Poliklinik für Orthopädie und Sportorthopädie, Klinikum rechts der Isar der TU München
- PD Dr. Sebastian Gehmert, Orthopädie, UKBB Universitäts-Kinderspital beider Basel
- Prof. Dr. Marcus Lehnhardt / Prof. Dr. Björn Behr, Klinik für Plastische Chirurgie und Handchirurgie, Sarkomzentrum Bergmannsheil Bochum, BG Universitätsklinikum Bergmannsheil
- Prof. Dr. Adrien Daigeler, Klinik für Hand-, Plastische, Rekonstruktive und Verbrennungschirurgie, BG Klinik Tübingen
Literatur
-
Kohl C, Aung T, Haerteis S, Papathemelis T. Assessment of breast cancer primary tumor material in a 3D in vivo model.Clin Hemorheol Microcirc. 2021;79(1):157-16
-
Drexler K, Schmidt KM, Jordan K, Federlin M, Milenkovic VM, Liebisch G, Artati A, Schmidl C, Madej G, Tokarz J, Cecil A, Jagla W, Haerteis S, Aung T, Wagner C, Kolodziejczyk M, Heinke S, Stanton EH, Schwertner B, Riegel D, Wetzel CH, Buchalla W, Proescholdt M, Klein CA, Berneburg M, Schlitt HJ, Brabletz T, Ziegler C, Parkinson EK, Gaumann A, Geissler EK, Adamski J, Haferkamp S, Mycielska ME. Cancer-associated cells release citrate to support tumour metastatic progression. Life Sci Alliance. 2021 Mar 23;4(6):e202000903.
-
Pion E, Asam C, Feder AL, Felthaus O, Heidekrueger PI, Prantl L, Haerteis S, Aung T. (2020). Laser speckle contrast analysis (LASCA) technology for the semiquantitative measurement of angiogenesis in in-ovo-tumor-model. Microvasc Res. 2020 Sep 16:104072. doi: 10.1016/j.mvr.2020.104072.
-
Troebs J, Asam C, Pion E, Prantl L, Aung T, Haerteis S (2020). 3D monitoring of tumor volume in an in vivo model. Clin Hemorheol Microcirc. 2020 Sep 8. doi: 10.3233/CH-209216.
-
Feder AL, Pion E, Troebs J, Lenze U, Prantl L, Htwe MM, Phyo A, Haerteis S, Aung T (2020). Extended analysis of intratumoral heterogeneity of primary osteosarcoma tissue using 3D-in-vivo-tumor-model. Clin Hemorheol Microcirc. 2020 Sep 7. doi: 10.3233/CH-209204.
-
Lamby P, Minkow A, Handt S, Falter J, Schellenberg EL, Graf S, Hiebl B, Haerteis S, Gemeinhardt O, Krüger-Genge A, Klosterhalfen B, Jung EM, Franke RP, Momeni A, Prantl L, Jung F (2020). Histological and SEM Assessment of Blood Stasis in Kidney Blood Vessels after Repeated Intra-Arterial Application of Radiographic Contrast Media. Life (Basel) 2020 Aug 27;10(9):E167. doi: 10.3390/life10090167.
-
Stolwijk JA, Sauer L, Ackermann K, Nassios A, Aung T, Haerteis S, Bäumner AJ, Wegener J, Schreml S (2020). pH sensing in skin tumors: Methods to study the involvement of GPCRs, acid-sensing ion channels and transient receptor potential vanilloid channels. Exp. Dermatol. 2020 Jul 13. doi: 10.1111/exd.14150.
-
Prantl L, Eigenberger A, Gehmert S, Haerteis S, Aung T, Rachel R, Jung EM, Felthaus O. J. Clin Med. 2020 May 26;9(6):E1616. Enhanced Resorption of Liposomal Packed Vitamin C Monitored by Ultrasound.
-
Asam C, Buerger K, Felthaus O, Brebant V, Rachel R, Prantl L, Witzgall R, Haerteis S, Aung T. Subcellular localization of the chemotherapeutic agent doxorubicin in renal epithelial cells and in tumor cells using correlative light and electron microscopy. Clin Hemorheol Microcirc. 2019 Sep 18. doi: 10.3233/CH-199212.
-
Aung T, Asam C, Haerteis S. Ion channels in sarcoma: pathophysiology and treatment options. Pflugers Arch. 2019 Sep;471(9):1163-1171.
-
Kather JN, Hörner C, Weis CA, Aung T, Vokuhl C, Weiss C, Scheer M, Marx A, Simon-Keller K (2019). CD163+ immune cell infiltrates and presence of CD54+ microvessels are prognostic markers for patients with embryonal rhabdomyosarcoma. Sci Rep. 25;9(1):9211.
-
Kauffmann P, Troeltzsch M, Brockmeyer P, Bohnenberger H, Heidekrüger PI, Manzke M, Canis M, Gaayathiri S, Schliephake H, Prantl L, Aung T (2018). First experience of chick chorioallantoic membrane (CAM) assay in the clinical work flow with oral squamous cell carcinoma patients. Clin Hemorheol Microcirc. 70:487-494.
- Kauffmann P, Troeltzsch M, Cordesmeyer R, Heidekrueger PI, Schliephake H, Canis M, Wolff HA, Rave-Fraenk M, Stroebel P, Kehrer A, Prantl L, Aung T (2017). Presentation of a variation of the chorioallantoic membrane set up as a potential modell for individual therapy for squamous cell carcinoma of the oropharynx. Clin Hemorheol Microcirc. 67:453-457.
- Sperling S, Aung T, Martin S, Rohde V, Ninkovic M (2017) Riluzole: a potential therapeutic intervention in human brain tumor stem-like cells. Oncotarget 8:96697-96709.
- Koch R*, Aung T*, Vogel D, Chapuy B, Wenzel D, Becker S, Sinzig U, Venkataramani V, von Mach T, Jacob R, Truemper L, Wulf GG (2016) Nuclear trapping through inhibition of exosomal export by indomethacin increases cytostatic efficacy of doxorubicin and pixantrone. Clin Cancer Res. 22:395-404.
- Koch R, Demant M, Aung T, Diering N, Cicholas A, Chapuy B, Wenzel D, Lahmann M, Güntsch A, Kiecke C, Becker S, Hupfeld T, Venkataramani V, Ziepert M, Opitz L, Klapper W, Trümper L, Wulf GG (2014). Populational equilibrium through exosome-mediated Wnt signaling in tumor progression of diffuse large B-cell lymphoma. Blood 12:2189-98.
AG Büchler - Adipositas und Lebererkrankungen
Leitung der Arbeitsgruppe:
- Prof. Dr. Christa Büchler
Mitglieder der Arbeitsgruppe:
- Susanne Feder
- Dr. Elisabeth Haberl
- Marlen Spirk
Chemerin ist ein Chemoattraktant für Immunzellen, es reguliert aber auch die Adipogenese und den Glukosestoffwechsel. Systemisches Chemerin wird vor allem durch dessen Synthese in Adipozyten bestimmt und ist bei Übergewicht erhöht. Im Serum findet sich zumeist inaktives Chemerin, welches durch verschiedene Proteasen prozessiert und aktiviert wird. Dies findet vorwiegend im Rahmen von Entzündungsprozessen statt. Übergewicht ist ein Risikofaktor für verschiedene Lebererkrankungen wie das hepatozelluläre Karzinom. Vom Fettgewebe freigesetzte Proteine, die sogenannten Adipokine, haben in diesem Zusammenhang eine große Bedeutung.
Die Arbeitsgruppe untersucht die Rolle von Chemerin im hepatozellulären Karzinom. Ziel ist es, die biologische Funktion der unterschiedlichen Chemerin Isoformen in Hepatozyten und hepatischen Sternzellen zu klären. Des Weiteren wird der Effekt einer hepatischen Chemerin Überexpression auf die Entstehung und Progression des hepatozellulären Karzinoms untersucht. Letztendlich wird die Expression von Chemerin und dessen Rezeptoren in humanen Geweben analysiert.
In Kooperation mit dem Institut für Klinische Chemie und Laboratoriumsmedizin wird untersucht inwieweit sich das Lipidom von Lebertumoren verändert. Dies gibt Hinweise auf den veränderten Stoffwechsel von Tumorzellen und somit mögliche Ansätze für deren Therapie. Weiterhin wird versucht systemische Biomarker für die Diagnose zu identifizieren.
Förderung
![]()
Stiftung für Pathobiochemie und Molekulare Diagnostik der Deutschen Vereinten Gesellschaft für Klinische Chemie und Laboratoriumsmedizin e.V.
AG Experimentelle Chirurgie
Leitung der Arbeitsgruppe:
- Prof. Edward K. Geissler, PhD & Prof. Dr. med. Hans J. Schlitt
Mitglieder der Arbeitsgruppe:
- Dr. med. Lucia Baier Kleinhenz
- Dr. med. Florian Bitterer
- Dr. med. Frank Brennfleck
- Prof. Dr. med. Stefan Brunner
- PD Dr. med. Dr. rer. nat. Axel Doenecke
- PD Dr. rer. nat. Elke Eggenhofer
- Dr. med. Markus Götz
- PD Dr. med. Christina Hackl
- Prof. Dr. med. Matthias Hornung
- Dr. med. James Hutchinson
- Dr. med. Henrik Junger
- Dr. rer. nat. Gudrun Köhl
- Dr. Katharina Kronenberg, PhD
- PD Dr. Maria Mycielska, PhD
- Dr. Paloma Riquelme, PhD
- Prof. Dr. med. Marcus Scherer
- Dr. med. Katharina Schmidt
- PD Dr. med. Jens Werner
- Dr. med. Daniel Wöhl
Wichtige Forschungsschwerpunkte der Klinik und Poliklinik für Chirurgie und der Experimentellen Chirurgie liegen in der Krebsgrundlagenforschung und der Entwicklung von Krebserkrankungen nach Transplantation. Immunsupprimierte Transplantat-empfänger sind besonders anfällig für Krebs, was eine häufige Nebenwirkung der Langzeitanwendung von Immunsuppressiva ist. Die Professoren Geissler und Schlitt haben die bislang größte internationale klinische Studie zu den Auswirkungen von Immunsuppression auf das Wiederauftreten von Hepatozellulären Karzinomen (HCC) bei Lebertransplantatempfängern durchgeführt (www.silver-study.org). Die verschiedenen Arbeitsgruppen der Abteilung untersuchen immunologische Aspekte der Krebsbiologie sowie Metabolomik und kritische Zellsignalwege, die die Entstehung von Krebs beeinflussen. Neben der Anwendung von Tierversuchsmodellen sind wir kontinuierlich an klinischen Studien beteiligt, und pflegen enge interdisziplinäre Kooperationen mit verschiedenen Forschergruppen am UKR um positive Fortschritte in der onkologischen Forschung zu erzielen. Die Klinik und Poliklinik für Chirurgie führt umfassende Grundlagenforschung durch, ist aber auch bestrebt, diese Forschungsergebnisse in klinische Studien zu übertragen, die wiederum zu besseren Behandlungsmöglichkeiten für Krebspatienten führen.
Forschungsgebiete
- Immunmonitoring
- Immuntherapie
- Molekulare Onkologie
- Metabolismus von Krebszellen
- Metronomische Therapie
- Tumorerkrankungen nach Transplantation
- Zelltherapie
Methoden
- Verschiedene Modelle der Tumorbildung bei Mäusen
- Intravitalmikroskopie
- System zur Sammlung humanen Gewebes (Tumor, Blut, etc.)
- Qualitätskontrolle und Immune Monitoring Labor (Dr. med. James Hutchinson)
- Krebsmigration, Angiogenese und Immune Assays
- Molekulare und Zellbiologische Methoden
- Zellisolation und -charakterisierung (MACS, Flow Cytometry)
Qualitätskontrolle (QK) und Immune Monitoring Labor
Die Qualitätskontrolle (QK) ist ein GMP- (Good Manufacturing Practice) zertifiziertes Analyselabor der Chirurgie/Experimentellen Chirurgie und Transfusionsmedizin. Das QK-Labor ist verantwortlich für die pharmazeutische Testung zur Freigabe von Transfusionsprodukten wie Thrombozyten, Granulozyten, CD34 + hämatopoetischen Stammzellen und extrakorporalen Photopheresaten. Darüber hinaus führt das QK-Labor eine Vielzahl von standardisierten Immunmonitoring-Assays durch, u.a. die umfassende Phänotypisierung peripherer Blutleukozyten und Tests der Leukozytenfunktion. Als Service für klinische Forscher am UKR führt das QK-Labor seine Standardtests auf Anfrage durch oder unterstützt klinische Studien. Bei entsprechender Nachfrage entwickelt und validiert das QK-Labor in Zusammenarbeit mit anderen Forschungsgruppen neue klinische Assays.
Verantwortliche Personen - Chirurgie:
- Chirurgie: Dr. med. James Hutchinson - Laborleiter (T: 6831)
- Dr. Katharina Kronenberg, PhD - Laboraufsicht (T: 4483 o. 6923)
Verantwortliche Personen - Transfusionsmedizin:
- PD Dr. Robert Offner - Sachkundige Person (T: 6266)
- Dr. rer. nat. Viola Hähnel - Laborleiterin (T: 9915)
Klinische Studiengruppe
Unser Studienteam bietet eine große Bandbreite an Dienstleistungen zur Unterstützung von klinischen Studien (v.a. IITs). Das Team besteht aus medizinischem Fachpersonal, Experten im Gesundheitswesen und Wissenschaftlern mit umfangreicher internationaler Erfahrung bei der Initiierung und Durchführung von klinischen Prüfungen der Phase I-IV, gemäß der ethischen Grundsätze der Deklaration von Helsinki, den nationalen Gesetzen (wie AMG, GCP-V) und internationalen Leitlinien (ICH-GCP). Unser Team kooperiert mit dem Zentrum für Klinische Studien (ZKS).
Laufende Forschungsprojekte
- Deutsche Krebshilfe (DKH) Förderschwerpunktprogramm "Translationale Onkologie": "Präkonditionierung von Tumor, Tumormikromilieu und dem Immunsystem für Immuntherapie (PROMIT Studie)" Projekt 4: "Understanding and predicting therapy outcomes by liquid biopsy", PD Dr. med. S. Haferkamp, Dermatologie, Prof. E. K. Geissler, PhD, Chirurgie, Prof. Dr. med. C. Klein, Experimentelle Medizin und Therapieverfahren
- DFG Projekt „Mechanismus der extrazellulären Citrataufnahme und Metabolismus bei Krebs; Spezifität und mögliche Verwendung von Gluconat in der Krebstherapie“, Prof. E. K. Geissler, PhD, und PD Dr. M. Mycielska, PhD
- DFG Projekte im Rahmen der Forschergruppe (FOR 2127) „Selektion und Adaptation während der metastatischen Krebsprogression“ (Prof. Dr. med. Christoph Klein, Koordinator) Teilprojekt B2: „Überwachung und Kontrolle metastasierender Tumorerkrankungen durch das angeborene Immunsystem“, Prof. E. K. Geissler, PhD, und Dr. med. P. Renner, Teilprojekt B3: „Die Bedeutung nicht-kodierender RNA in Metastasierungs-prozessen und Therapieresistenz“, PD Dr. med. C. Hackl, Prof. Dr. G. Meister
- Else Kröner-Fresenius-Stiftung "HCCIS Study: Prospective, Multicenter Study to Evaluate the Hepatocellular Carcinoma Immune Score as Prognosticator for Overall and Disease-free Survival of Patients After Resection of Hepatocellular Carcinoma", Prof. Dr. med. S. Brunner
- „Else-Kröner-Forschungskolleg Regensburg – Interdisziplinäre translationale Immuno-Onkologie Forschungsprogramm“, Dr. med. F. Bitterer
- Else Kröner-Fresenius-Stiftung, “Der plasmamembranständige Citrattransporter pmCIC im humanen Pankreaskarzinom und seine Bedeutung für Tumorproliferation und Metastasierung" Dr. med. K. Schmidt
- "virtual Global Transplantation Laboratory (vGTL)" Initiative sponsored by the TTS with The ONE Study to develop international standardized protocols for immunological and genetic monitoring of human transplant recipients, including ONE Study cell phenotyping panels now marketed by Beckman Coulter Life Sciences, Prof. E. K. Geissler , PhD
- Bristol-Myers Squibb Stiftung “Virus-spezifische Gedächtnis-T-Zell-Reaktionen, die durch die Blockade des Immun-Checkpoints demaskiert werden, verursachen Hepatitis” Dr. med. J. Hutchinson, PD Dr. med. S. Haferkamp, Dermatologie, Prof. E. K. Geissler, PhD
- Bristol-Myers Squibb Stiftung “Immune Monitoring of Checkpoint Inhibitor Therapy in Melanoma” Dr. med. J. Hutchinson, PD Dr. med. S. Haferkamp, Dermatologie, Prof. E. K. Geissler, PhD
AG Experimentelle, gynäkologische Onkologie (Klinik für Frauenheilkunde und Geburtshilfe)
Leitung der Projekte:
- Prof. Dr. rer. nat. Gero Brockhoff
- Prof. Dr. med. vet. Anja Kathrin Wege
Die Onkologische Arbeitsgruppe „Experimentelle, gynäkologische Onkologie“ am Lehrstuhl für Frauenheilkunde und Geburtshilfe (Schwerpunkt Frauenheilkunde) am Caritaskrankenhaus St. Josef der Universität Regensburg setzt sich aus zwei eng kooperierenden Untergruppen zusammen, einmal die Zytometrische und Zelluläre Onkologie (Brockhoff) und die Experimentelle Immunologie (Wege) (Homepage der Arbeitsgruppe). Die AG beschäftigt sich mit der Geno- und Phänotypisierungen, sowie der funktionellen Charakterisierung (z. B. ErbB-Rezeptor-Tyrosin-Kinasen) von gynäkologischen Malignomen (Schwerpunkt Mammakarzinom) und den sich daraus ergebenden Antigen-spezifischen Therapien. Darüber hinaus stehen Mechanismen einer immunologischen Tumorabwehr bzw. der Einsatz neuartiger Immuntherapien im Fokus, die darauf abzielen, die körpereigene Immunantwort auf malignes Zellwachstum zu stimulieren. Das Spektrum der Untersuchungen basiert auf in-vitro (versch. 2D und 3D Zellkulturmodelle), ex-vivo (primäres Tumormaterial von Mammakarzinompatientinnen) und in-vivo (tierexperimentellen) Untersuchungen. Der Einsatz von Humanisierten Tumormäusen (HTM) für die translationale Evaluation von Antigen-spezifischen oder Immuntherapien ist in der AG von zentraler Bedeutung. HTM zeichnen sich durch die Koexistenz von humanem Tumorwachstum und einem humanen Immunsystem aus und kommen von daher der Situation im Patienten relativ nahe. Insofern können im HTM Modell klinisch relevante Fragestellungen sehr präzise adressiert werden und Therapiestudien mit großer Aussagekraft durchgeführt werden.
www.caritasstjosef.de/frauenheilkunde-geburtshilfe/forschung
AG Experimentelle Tumorpathologie

Leitung der Arbeitsgruppe:
- Prof. Dr. med. Diego F. Calvisi
Mitglieder der Arbeitsgruppe:
-
Prof. Dr. med. Matthias Evert
-
Dr. rer. nat. Antonio Cigliano
-
Dr. med. Katja Evert
-
Dr. med. Kirsten Utpatel
-
Alexander Scheiter, Assistenzarzt
-
Manfred Meyer, MTA
-
Ingrid Winkel, MTA
Die Arbeitsgruppe für Experimentelle Tumorpathologie gehört zum Institut für Pathologie der Universität Regensburg. Hauptfokus unserer Forschung ist die Aufdeckung molekularer Mechanismen, die die Entstehung und Progression von Lebertumoren verursachen. Darauf aufbauend wollen wir innovative Therapiestrategien gegen diese Tumoren entwickeln.
Leberkrebs ist weltweit der fünfthäufigste Tumor und die zweithäufigste krebsbedingte Todesursache überhaupt. Beim Erwachsenen ist das hepatozelluläre Karzinom (HCC) der häufigste Tumor, gefolgt vom cholangiozellulären Karzinom (Cholangiokarzinom, Gallengangskarzinom, CCA). Das Hepatoblastom ist der häufigste maligne Lebertumor des Kindesalters. Unabhängig vom histologischen Typ sind alle genannten Lebertumoren hoch aggressiv. Es gibt nur eine beschränkte Möglichkeit der Therapie, wenn - was leider oft der Fall ist - eine chirurgische Entfernung des Tumors nicht mehr möglich ist. Dies liegt vor allem daran, dass wir über die molekularen Ursachen in der Entstehung dieser Tumoren bisher viel zu wenig wissen.
Genau hier setzen wir an. Unsere experimentellen Ansätze sind vielseitig. Wir benutzen eine Reihe verschiedener in vitro- und in vivo-Modelle, die wir stetig weiterentwickeln. Wir fokussieren uns dabei insbesondere auf die Bedeutung verschiedener Signalwege, z.B. AKT/mTOR, Ras/MAPK, Hippo, Notch, Wnt/Beta-catenin, Retinoblastoma/E2F1. Nicht nur die Aktivität einzelner Signalwege, sondern insbesondere deren funktionelles Zusammenspiel steht dabei im Vordergrund unserer Untersuchungen, sowohl bei der Tumorentstehung als auch bei der Tumorprogression. Insbesondere richten wir unsere Aufmerksamkeit darauf, das Tumorwachstum durch innovative, molekulare und genau auf diese Signalwege gerichtete Therapien hin zu hemmen. Wir sind davon überzeugt, dass wir auf diese Weise neue Wege aufdecken können, die es ermöglichen, Tumorzellen zu eliminieren oder wenigstens substantiell das Tumorwachstum in einer Weise zu verzögern, wie es bisher nicht möglich ist.
Besonderes Interesse gilt zudem dem Tumormetabolismus, speziell bei der malignen Transformation von Leberzellen aber auch während des Tumorwachstums und der Tumorprogression. Es gibt immer mehr Beweise dafür, dass die Fähigkeit von Tumorzellen, ihren Zellmetabolismus zu reprogrammieren eine entscheidende Voraussetzung für das Tumorwachstum ist ("Hallmarks of Cancer"). In diesem Zusammenhang untersuchen wir u.a. den Lipidmetabolismus von Tumorzellen, konkret die aberrante de novo-Lipidsynthese und die Cholesterol-Biosynthese. Dabei wollen wir wiederum speziell die molekularen Mechanismen identifizieren, die es den Karzinomzellen ermöglichen, den Lipidmetabolismus so umzustellen, dass sie resistent auf einen Entzug von freien Fettsäuren reagieren. Hieraus könnten sich interessante Therapieansätze, z.B. durch Kombination von Inhibitoren der Fettsäuresynthese mit konventionellen oder molekularen Therapiestrategien, ergeben, da die Toxizität eines einzelnen Wirkstoffs durch Dosisreduktion verringert wird. Speziell diese Aspekte sind nicht spezifisch für Krebszellen der Leber sondern in vielen Tumoren relevant, so dass Ergebnisse unserer Forschung breitere Bedeutung erlangen können.
Onkogene sind krebsfördernde Gene und Tumorsuppressoren schützen vor der Tumorentwicklung. Über diese allgemeinen Grundsätze hinaus ist aber relativ wenig Konkretes darüber bekannt, wie sich multiple Veränderungen in Onkogenen und Tumorsuppressoren - die in Tumoren des Menschen oft synchron auftreten - tatsächlich gegenseitig beeinflussen und welche Auswirkungen dies auf die Tumorentwicklung und auf die Auswahl von Therapiestrategien haben kann. Um hier neue Kenntnisse zu erzielen, nutzen wir eine Technik zur Erzeugung von Lebertumoren in der Maus, die "Hydrodynamischer Gentransfer" genannt wird. Sie basiert auf der Injektion einer großen Menge DNA in die Schwanzvene, die die Gene, die es zu untersuchen gilt, beinhaltet. Gleichzeitig wird das Gen der Sleeping-Beauty-Transposase co-injiziert, so dass die Gene vor allem in Hepatozyten in der Leber stabil überexprimiert werden. Auf diese Weise haben wir in Zusammenarbeit mit kooperierenden Arbeitsgruppen auf der ganzen Welt wegweisende und zukunftsträchtige Entdeckungen gemacht, die den Kenntnisstand zur Entstehung von Lebertumoren, der Tumorzelldifferenzierung, der Signaltransduktion, der Plastizität einer Zelldifferenzierung und die Resistenzentwicklung gegenüber Therapien bereichert haben. Dies liegt auch daran, dass wir die im Mausmodell enhaltenen Ergebnisse stets in Zellinien aus menschlichen Tumoren und in einer großen Kollektion von Tumorgewebe des Menschen validieren konnten.
Bedingt durch die große Expertise am Standort Regensburg beginnen wir nun auch, die Bedeutung des Mikromilieus des Tumors und den Einfluss des Immunsystems in Lebertumoren durch verschiedene experimentelle Modelle und in den Tumoren des Menschen zu untersuchen.
AG Experimentelle Urologie
Leitung der Arbeitsgruppe:
- Prof. Dr. med. Maximilian Burger (Ordinarius)
- PD Dr. med. Wolfgang Otto (Forschungskoordinator, Laborleiter)
Mitglieder der Arbeitsgruppe:
-
Dr. med. Johannes Breyer (Doktorandenbeauftragter)
-
Dr. med. Johannes Bründl (Stv. Laborleiter)
-
Dr. med. Philipp Spachmann
-
Dr. rer. nat. Sabine Wallinger
-
Dipl.-Biochemikerin Patricia Luckner
-
MTA Stefanie Götz
-
MTA Nina Niessl
-
MTA Linda Otzelberger
Urologisches Forschungslabor
Neben den Aufgaben als urologische Klinik der Maximalversorgung sowie der studentischen Ausbildung wird am Lehrstuhl für Urologie der Universität Regensburg auch der laborexperimentellen Forschungsarbeit eine besondere Bedeutung zugemessen. Die Bedeutung des Regensburger Lehrstuhls in der urologischen Forschungslandschaft zeigt sich neben der herausragenden Publikationsleistung der vergangenen Jahre nicht zuletzt daran, dass Ordinarius Prof. Dr. med. Maximilian Burger dem Ressort Forschungsförderung der Deutschen Gesellschaft für Urologie vorsteht.
Wissenschaftliche Schwerpunkte
Die wissenschaftlichen Schwerpunkte des urologischen Forschungslabors liegen traditionell in der Erforschung von (molekularen) Progressionsmarkern zur Charakterisierung des Harnblasenkarzinoms unter besonderer Beachtung des frühinvasiven Tumorstadiums. Weitere Schwerpunkte der Forschung sind das Prostata-, Nieren- und Peniskarzinom, außerdem die topographische Anatomie der Prostata. Darüber hinaus werden in Kooperation mit der dem Lehrstuhl für Urologie angeschlossenen Klinik für Kinderurologie (Leitung: Prof. Dr. med. Wolfgang Rösch) wissenschaftliche Fragestellungen aus dem Bereich der Kinderurologie untersucht.
Kooperationspartner
Seit dem Jahr 2012 stehen der Klinik und Poliklinik für Urologie für die experimentelle Forschungsarbeit vergrößerte und modernisierte Räumlichkeiten im Forschungsbau H4 am Uniklinikum Regensburg zur Verfügung. Es bestehen zahlreiche Kooperationen mit Forschungsgruppen im In- und Ausland. Dazu gehören u.a. die urologischen Universitätsabteilungen in Bozen (I), Erlangen, Leipzig, Mainz, Mannheim, Prag (CZ), Ufa (RUS), Ulm und Würzburg sowie die pathologischen Institute der Universitätskliniken Regensburg, Erlangen, Frankfurt und Zürich (CH) sowie die Jack-Burch-Unit der University of York (UK) und das Labor der STRATIFYER Molecular Pathology GmbH Köln.
AG Garcke - Mathematical modeling of tumor growth
Leitung der Arbeitsgruppe:
- Prof. Dr. Harald Garcke
Mitglieder der Arbeitsgruppe:
- PD Dr. Anca Matioc
- PD Dr. Bogdan Matioc
- Dr. Patrik Knopf
- Matthias Ebenbeck
Die Arbeitsgruppe beschäfigt sich mit der quantitativen Beschreibung von räumlichen Tumorwachstum. In den letzten 10 Jahren sind sowohl mathematische Beschreibungen die auf Probleme mit freiem Rand als auch Phasenfeldmodelle entwickelt und analysiert worden. Dabei können Aspekte wie zum Beispiel der Einfluss von Nährstoffen und Medikamenten, Vaskularisierung, Angiogenesis und Nekrosis berücksichtigt werden. Mittels Stabilitätsanalyse konnte der Übergang von einem kompakten Wachstum zu einem verzweigten Wachstum in Abhängigkeit von Parametern nachgewiesen werden. Mit Hilfe von Optimierungsmethoden hat die Arbeitsgruppe Fragestellungen wie die optimale Berechnung von Behandlungszeiten bearbeitet. Aktuell werden Methoden entwickelt um auf der Basis von patientenspezifischen Daten Parameter aus Daten zu lernen bzw. zu identifizieren.
Methoden
- Ortsaufgelöste Kontinuumsmodellierung von Tumorwachstum
- Computersimulation mit Finiten Elementen
- Optimierung mit partiellen Differentialgleichungen als Nebenbedingungen zur Behandlungsoptimierung
- Lernen von Parametern mittels Proper Orthogonal Decomposition
- Stabilitätsanalyse der Parameterabhängigkeit von Tumorwachstum
AG Interaktion von Anästhetika und Analgetika mit Tumorzellen in vitro
Leitung der Arbeitsgruppe:
- PD Dr. Anika Bundscherer
Es gibt Hinweise darauf, dass die perioperative Phase eine vulnerable Phase darstellt, in der Metastasierung und Tumorprogession begünstigt werden können. Die Kombination aus chirurgischer Manipulation und perioperativer Kompromittierung der Immunkompetenz erhöht das Risiko einer Tumordissemination und könnte sich somit negativ auf den Krankheitsverlauf onkologischer Patienten auswirken. In diesem Zusammenhang stellt sich die Frage, welchen Einfluss die perioperativ eingesetzten Analgetika und Anästhetika auf die Tumorprogression haben. Im Rahmen der perioperativen Schmerztherapie werden üblicherweise Kombinationen aus Opioidanalgetika und Nicht- Opioidanalgetika wie Metamizol, Paracetamol und nicht steroidaleAntirheumatika (NSAR) angewandt. Bei einer Vielzahl von operativen Eingriffen wie abdominal- oder thoraxchirurgischen Operationen sowie Eingriffen an den Extremitäten kommen zusätzlich Regionalanästhesieverfahren zum Einsatz. Ziel dieses Projektes ist es, den Einfluss unterschiedlicher Anästhetika und Analgetika auf die Progression maligner Zellen unterschiedlicher Entität in vitro zu untersuchen.
AG Kardio-Onkologie
Leitung der Arbeitsgruppe:
- PD Dr. med. Can M. Sag
Mitglieder der Arbeitsgruppe:
- Dr. med. Daniel Tarnowski
- Klaus-Georg Kreitmeier (Arzt)
- Felicia Radtke (BTA)
- Magdalena Erhardt (cand. med.)
- Frederic Hachmeister (cand. med.)
- Thomas Sowa (BTA, AG Prof. Maier)
Die onkologische Substanzklasse der Anthrazykline führt bei bis zu 9% aller Patienten, die wegen eines Krebsleidens mit einer Anthrazyklin-haltigen Chemotherapie behandelt werden, zu einer Abnahme der systolischen Kontraktionskraft des Herzens. Klinisch resultiert eine sog. toxische Kardiomyopathie, die nur schwer behandelbar ist und die Langzeitprognose von Krebspatienten trotz überlebtem Krebsleiden signifikant verschlechtert.
Trotz der hohen Prävalenz und schlechten Prognose der toxischen Kardiomyopathie gibt es keine leitlinienbasierten Strategien zur Prävention oder Therapie dieser Form der Herzinsuffizienz, was u.a. auch daran liegt, dass die der toxischen Kardiomyopathie zugrundeliegenden Pathomechansimen auf zellulärer Ebene noch unzureichend verstanden sind.
Ziel unserer interdisziplinären Arbeitsgruppe Kardio-Onkologie ist es daher diejenigen Pathomechanismen zu erforschen, die auf zellulärer Ebene die Kardiotoxizität medikamentöser Tumortherapie am Herzen vermitteln.
Unser besonderer Arbeitsschwerpunkt liegt dabei auf der Charakterisierung der pathophysiologischen Bedeutung sog. radikaler Sauerstoffspezies (ROS) in der Herzmuskelzelle, deren Menge infolge einer Anthrazyklin-Therapie am Herzen krankhaft gesteigert ist. So könnten radikale Sauerstoffspezies durch Aktivierung redox-abhängiger Proteinkinasen wie der CaMKII wahrscheinlich entscheidend zum Krankheitsprogress der toxischen Kardiomyopathie beitragen, sodass wir im Verlauf unter Einsatz verschiedener transgener Mausmodelle untersuchen wollen, ob eine therapeutische Beeinflussung des CaMKII-abhängigen Signalwegs für eine Therapie der toxischen Kardiomyopathie geeignet sein könnte.
Etablierte Methoden
- Herzmuskelzellisolation (auch aus humanem Gewebe)
- Verschiedene fluoreszenzmikroskopische Techniken zur Untersuchung des intrazellulären Kalzium- und ROS-Stoffwechsels (u.a. Epifluoreszenzmikroskopie und konfokale Mikroskopie)
- Muskelstreifenexperimente
- Molekularbiologie
- Histologie
- Kleintierechokardiographie (u.a. Maus und Ratte)
- Kleintierchirurgie (u.a. transverse Aortenkonstriktion)
Aktuelle Forschungsförderung
- DFG-Sachbeihilfe SA 3282/1-1: „Die Bedeutung der cAMP- und redox-aktivierten Proteinkinase A (PKA) während pathologischer Nachlasterhöhung am Herzen“.
- Fördermittel im Rahmen der Regensburger Forschungsförderung in der Medizin (ReForM) Baustein A – Anschubfinanzierung: „Die Inhibition der Bruton Tyrosin Kinase schädigt den intrazellulären Kalziumstoffwechsel der Herzmuskelzelle“.
AG Klinische Psychologie und Psychotherapie
Leitung der Arbeitsgruppe:
- Prof. Dr. Andreas Mühlberger
Mitglieder der Arbeitsgruppe:
-
Dr. Stefanie Biehl
-
Benedikt Schröder
-
Melissa Schmidmeier
Die Arbeitsgruppe für Klinische Psychologie und Psychotherapie erforscht translational und prozessbezogen die emotionalen und verhaltensbezogenen Faktoren, die psychische Gesundheit und psychische und körperliche Erkrankungen beeinflussen. Dazu verwenden wir auch Virtuelle Realität, in der z.B. Expositionsübungen und Verhaltenstest bei Angststörungen sehr erfolgreich durchgeführt werden können. Außerdem erfassen wir das Befinden von Personen multidimensional unter Einbezug des Erlebens, der physiologischen Reaktionen und des Verhaltens.
Methoden
Virtuelle Realität (VR)
... erlaubt es dem Nutzer, sich in einer computergenerierten Umgebung zu bewegen und mit dieser zu interagieren. Die rasante Entwicklung der technischen Möglichkeiten für VR (z.B. bei Head Mounted Displays, HMDs bzw. VR Brillen) ermöglichen den breiten Einsatz immersiver VR. Es konnte bereits belegt werden, dass Expositionsübungen bei Ängsten in VR sehr erfolgreich durchgeführt werden können. Am Lehrstuhl stehen sowohl eine CAVE (5 Seiten Stereoprojektionssystem) als auch VR Brillen sowie Software für entsprechende Anwendungen zur Verfügung.
Multimodale Emotionserfassung
Bei unseren Untersuchungen wird das Befinden der Teilnehmer nach Möglichkeit multimodal erfasst. Neben dem Selbstreport durch Ratingfragen oder Fragebögen werden peripher-physiologische Reaktionen (z.B. Herzrate, Atmung, Hautleitfähigkeit) und kortikale Maße (EEG) erhoben sowie Verhalten und Aufmerksamkeitsprozesse über die Erfassung von Augen-und Körperbewegungen berücksichtigt. Die kontinuierlich erhobenen Daten können dann auch für Feedbackanwendungen genutzt werden.
Online Datenerhebung
Es besteht auch die Möglichkeit zur sicheren Online-Erfassung von Daten, insbesondere zur Erhebung von Daten in relevanten lebensnahen Situationen und in der Versorgungsforschung.
Laufende Forschungsprojekte
-
ViReTa (gefördert von der Deutschen Krebshilfe e.V.):
Ein wichtiger Risikofaktor für Krebserkrankungen ist Rauchen. Das Rauchen aufzugeben ist für viele Personen eine große Herausforderung und die Rückfallquoten nach einem erfolgreichen Rauchstopp sind immer noch sehr hoch. Deshalb führen wir gerade zusammen mit dem Universitätsklinikum für Psychiatrie und Psychotherapie der Universität Tübingen eine randomisierte, kontrollierte Studie an zwei Standorten (Tübingen, Regensburg) durch. In dieser vergleichen wir den Zusatznutzen eines Reizexpositions- und Ablehnungstrainings in Virtueller Realität als Add-on zu einem verhaltenstherapeutischen Raucherentwöhnungsprogramm mit dem eines zusätzlichen Entspannungstrainings.
-
Comptune (industriegefördert):
Die Compliance der Patienten ist ein wichtiger Faktor bei dem Erfolg sowohl von somatischen als auch psychotherapeutischen Interventionen. Gerade bei der Therapie von Glioblastomen mit Tumortherapiefeldern (TTFields) ist die Compliance eine wesentliche Komponente des Therapieerfolgs. In einem Kooperationsprojekt unter Leitung von Prof. Dr. Martin Pröschold untersuchen wir deshalb prognostische und prozessbezogene Faktoren, die die Compliance beeinflussen, um darauf aufbauend auch Interventionsstrategien zur Verbesserung der Compliance, sowohl spezifisch für die Therapie mit TTFields als auch allgemein für somatische und psychotherapeutische Interventionen ableiten zu können.
AG Kretz - Funktion langer nicht-kodierender RNAs in der Entwicklung von Hauttumoren
Leitung der Arbeitsgruppe:
- PD Dr. Markus Kretz
Markus Kretz gründete seine Arbeitsgruppe im Juni 2012 in Regensburg. Fokus der Forschung in der AG Kretz ist die Untersuchung des Wirkungsmechanismus von langen, nicht Protein-kodierenden RNAs in der Tumorentwicklung humaner Gewebe.
Während bisher nur wenige der annotierten langen nicht-kodierenden RNAs (lncRNAs) im Detail untersucht wurden, weisen bisherige Daten auf eine erstaunliche funktionelle Vielfalt in vielen biologischen Prozessen hin. Entsprechend spielen lncRNAs eine wichtige Rolle in der Entwicklung diverser Tumore.
Ein Forschungsschwerpunkt der AG Kretz liegt in der Untersuchung der Funktionen von lncRNAs im Plattenepithel-Karzinom, einem der drei häufigsten menschlichen Hauttumore.
Mit Hilfe von humanen, organotypischen Haut-Modellsystemen wird analysiert wie lncRNAs das homöostatische Gleichgewicht zwischen Stammzellen und hoch-differenzierten Zellen der Epidermis regulieren und welchen Einfluss Plattenepithelkarzinom-assoziierte lncRNAs in der Störung dieser Balance während der Tumorbildung haben.
www.ur.de/biologie-vorklinische-medizin/long-noncoding-rnas/
AG Längst - Molekulare Mechanismen der Epigenetik
Leitung der Arbeitsgruppe:
- Prof. Dr. Gernot Längst
Mitglieder der Arbeitsgruppe:
- Dr. Helen Hoffmeister
- Dr. Rodrigo Maldonado
- Elisabeth Silberhorn
- Regina Gröbner-Ferreira
- Simon Holzinger
- Sara Wernig-Zorc
- Carolina Rejas
Die Modifikation epigentischer Signale in Tumorzellen ist ein vielversprechender therapeutischer Ansatz. Um ihn in die Tat umzusetzen, braucht es tiefere Einsichten in die Epigenetik von Tumoren. Die AG Molekulare Mechanismen der Epigenetik untersucht die Wechselwirkung zwischen den DNA verpackenden Histonproteinen und den Chromatin modifizieren Enzymen die die Zugänglichkeit der DNA für alle DNA abhängigen Prozesse, wie Transkription und DNA Reparatur kontrollieren. Es zeigt sich, dass Chromatin modifizierende Maschinen eine übergeordnete Regulationsebene der Genexpression darstellen und in der überwiegenden Anzahl von Tumoren Proteine dieser Funktionsgruppe mutiert sind.
Die Kenntnis von den Wirkorten dieser Chromatin modifizierenden Komplexe im Genom, der Art und Weise, wie sie den genomischen oder epigenomischen Code interpretieren und die Zugänglichkeit der DNA regulieren gibt Aufschlüsse über die Mechanismen der Entwicklung vielzelliger Organismen und Prozessen der Zellentartung. Es sind bereits vielversprechende Substanzen (z.B. Deme- thylierungsagentien, Histondeacetylaseinhibitoren) für die Tumortherapie zugelassen, die epigenetische Signale modifizieren. Wir verwenden eine Kombination von biochemischen, biophysikalischen, zellbiologischen und bioinformatischen Ansätzen um die Auswirkungen der in Krebs mutierten Enzyme zu untersuchen und zielgerichtete Therapeutika zu entwickeln.
Methoden
- Bioinformatikanalyse der Transkriptome, Zellkernarchitektur und Chromatinorganisation von Zellen
- Mechanistische und strukturelle Analyse der Chromatin modifizierenden Maschinen
- Biochemische Rekonstruktion komplexer molekularer Proteinkomplexe
- Biophysikalische Quantifizierung von molekularen Interaktionen
Laufende Forschungsprojekte
- DFG Projekte im Rahmen der Sonderforschingsbereiches (SFB960) „RNP biogenesis: assembly of ribosomes and non-ribosomal RNPs and control of their function“.
- BMBF Forschungsprojekt „NeuroChrom - Global chromatin remodeling alters the gene expression profile in a neurodegenerative disease“
AG Leitzmann - Epidemiologie und Präventivmedizin
Leitung der Arbeitsgruppe:
- Prof. Dr. Dr. Michael Leitzmann
Mitglieder der Arbeitsgruppe:
- Dr. Beate Fischer
- Dr. Christine Welker
- Dr. Carmen Jochem
- Dr. Hansjörg Baurecht
- Andrea Hillreiner
- Anja Sedlmeier
- Tatjana Nazarenus
- Christine Töpfer
Michael Leitzmann leitet seit 2008 als Direktor das Institut für Epidemiologie und Präventivmedizin an der Universität Regensburg. Sein Forschungsschwerpunkt liegt in der Untersuchung von Zusammenhängen zwischen Ernährung, Körpermasse, körperlicher Aktivität und der Entwicklung von chronischen Erkrankungen, insbesondere maligner Tumore. Dies umfasst (i) methodische Forschung in Bezug auf die Erfassung körperlicher Aktivität und ihre individuellen Komponenten in groß angelegten epidemiologischen Studien; (ii) evidenzbasierter Erforschung von (kausalen) Zusammenhängen zwischen Lebensstilfaktoren und Tumorentitäten mit Hilfe von systematischen Reviews, Umbrella-Reviews und Mendelian Randomization Studien; und (iii) Untersuchung der individuellen und kombinierten Beziehung von Ernährung, Körpermasse, körperlicher Aktivität und Tumorrisiko, -progression, Überlebenszeit und Therapieansprechen.
Etablierte Methoden
- Epidemiologische Studien
- Systematische Reviews
- Umbrella-Reviews
- Mendelian Randomization (Causal Effect Estimation)
Laufende Forschungsprojekte
- NAKO-Gesundheitsstudie (01ER1301/19) finanziert aus Mitteln des Bundes, der beteiligten Länder sowie der beteiligten Helmholtz-Zentren (www.nako.de)
- G-BA: INTEGRATION-Programm (01INVF18018) “Kombinierte Ernährungs- und körperliche Aktivitätsintervention während einer onkologischen Therapie“ in Kooperation mit PD. Freerk T. Baumann, Uniklinik Köln & Prof. Sebastian Theurich, Universitätsklinikum München
- Deutsche Krebshilfe: STOP-Studie (70112048) “Der präventive Effekt von Sensomotorik- und Vibrationstraining auf Oxaliplatin- und Vinca-alkaloid-induzierter Polyneuropathie - STOP“ in Kooperation mit PD. Fiona Streckmann, Deutsche Sporthochschule Köln
- AG-Mendelian Randomization Studies in Kooperation mit PD. Sebastian E. Baumeister, UNIKA-T Augsburg
AG Neurologische NeuroOnkologie
Leitung der Arbeitsgruppe:
- Prof. Dr. med. Peter Hau
Mitglieder der Arbeitsgruppe:
-
Dr. med. Tareq Haedenkamp
-
Dr. Elisabeth Bumes
-
Dr. Tareq Haedenkamp
-
Anna Fischl
-
Dr. Antje Kretner
-
Dr. Anna Pyschkin
-
Dr. Katrin Scheu
-
Dr. Edith Vandieken
-
Sonja Lugauer
-
Birgit Hirschmann
-
Lena Kaiser
-
Heike Weißwange
-
Dr. rer. nat. Arabel Vollmann-Zwerenz
-
Birgitt Jachnik
- Laura Marie Ammer
Die Arbeitsgruppe neurologische NeuroOnkologie beschäftigt sich unter grundlagenwissenschaftlichen, translationalen und klinischen Aspekten mit der Biologie von Tumorzellen des zentralen Nervensystems (ZNS). Es bestehe enge Kooperationen mit den Kliniken für Neurochirurgie, Medizinische Klinik III (Onkologie) und Strahlentherapie, mit den Abteilungen für Neuropathologie und Nuklearmedizin, dem Zentrum für Neuroradiologie sowie weiteren Partnern.
Die einzelnen Projekte beschäftigen sich mit klinischen Fragestellungen, mit der Frage nach den Mechanismen, die die Entstehung und Weiterentwicklung maligner Hirntumoren ermöglichen, sowie mit der Suche nach neuen therapeutischen Ansätzen in der kliniknahen Grundlagenforschung.
In der Grundlagenforschung werden mehrere Schwerpunkte mit Fokus auf Tumorzellinvasion (in Kooperation mit dem Lehrstuhl für Experimentelle Medizin und dem Fraunhofer-Institut), Tumormetabolismus (in Kooperation mit der Klinik und Poliklinik Innre Medizin III des UKR und dem Institut für Funktionelle Genomik der UR) und Interaktion von Tumorzellen mit dem Immunsystem (in Kooperation mit dem RCI) bearbeitet.
AG Neuropathologie
Leitung der Arbeitsgruppe:
Mitglieder der Arbeitsgruppe:
Als einzige Einrichtung ihrer Art gewährleistet die Abteilung für Neuropathologie des Universitätsklinikum Regensburg die neuropathologische Versorgung des gesamten ostbayerischen Raumes. Demzufolge steht ein breit gefächertes neuropathologisches Untersuchungsgut zur Verfügung, das in Kooperation mit unseren klinischen Partnern im Rahmen innovativer diagnostischer und therapeutischer Ansätze und translationaler wissenschaftlicher Aspekte analysiert wird.
Der Neuropathologie ist ein wichtiger Lotse in der modernen Tumortherapie. Mittels neuer Verfahren der Hochdurchsatz-Sequenzierung, die auch unter dem Begriff Next-Generation Sequencing (NGS) bekannt sind, können wir auch aus kleinsten Tumorgewebsproben Nukleinsäuren für eine molekulare Diagnostik und parallele Analyse zahlreicher tumorrelevanter Gene gewinnen. Hiermit ist dann eine sehr exakte neuropathologische Diagnosestellung sowie eine Vorhersage des Therapieansprechens möglich, die die Behandlungsqualität von Hirntumorpatienten am CCCO verbessert.
Im wissenschaftlichen Kontext erlaubt die Korrelation der erhobenen molekularen Befunde mit klinischen Daten zu Überlebenszeit und Therapieansprechen die Identifikation neuer diagnostischer, prognostischer und prädiktiver Biomarker. Ganz besonders interessieren uns hierbei die molekularen Veränderungen in bösartigen Hirntumoren (malignen Gliomen bzw. Glioblastomen). Ein wichtiger Qualitätsindikator unserer Forschung ist die in unserer Abteilung vorhandene Gewebsexpertise. So werden die erhobenen molekularen Befunde immer im Kontext der zugrundeliegenden Histologie betrachtet. Dies erlaubt eine gezieltere Interpretation, da unterschiedlichen Tumorarten (Entitäten) oder z.B. auch der intratumoralen Heterogenität im Glioblastom Rechnung getragen werden kann. Abgerundet wird unsere Forschung durch Untersuchungen in entsprechenden in vitro (Glioblastom-Stammzellen) und in vivo-Modellen, in denen wir neue Glioblastom-relevante Kandidatengene weiter funktionell charakterisieren.
Unsere Forschung wir extern durch zahlreiche Drittmittelgeber finanziert (z.B. Deutsche Krebshilfe, Mercator-Stiftung, Sibylle Assmus-Stiftung, Sander-Stiftung, Bayerisches Staatsministerium für Wissenschaft und Kunst, Else Kröner-Fresenius-Stiftung). Die Abteilung für Neuropathologie ist hierbei in Regensburger und überregionale Forschungsverbünde eingebunden.
AG Pädiatrische und Molekulare Onkologie
Leitung der Arbeitsgruppe:
- Dr. rer. nat. Gunhild Sommer
Mitglieder der Arbeitsgruppe:
- Dr. rer. nat. Tilman Heise (Projektleiter)
- Marie Matthes, M.Sc.
- Christina Sendlmeier, M.Sc.
- Rebecca Wätzig, B.Sc.
Das Ziel der Arbeitsgruppe Pädiatrische und Molekulare Onkologie der Abteilung Pädiatrische Hämatologie, Onkologie und Stammzelltransplantation (PHOS) ist es, wissenschaftliche Erkenntnisse aus der molekularen Forschung in moderne und nebenwirkungsarme Therapiekonzepte für bösartige Tumorerkrankungen im Kindesalter umzusetzen. Die Abteilung PHOS wird von Prof. Dr. Selim Corbacioglu geleitet.
Forschungsprojekte
(1) RIST-Therapie: Zur Behandlung von Patienten mit rezidiviertem und therapierefraktärem Neuroblastom wurde in unserer Abteilung ein neuartiger multimodaler Therapieansatz entwickelt, in dem molekular wirksame Inhibitoren bekannter Signalwege (Rapamycin, Dasatinib) mit konventioneller Chemotherapie (Irinotecan, Temozolomide) in metronomischer Weise kombiniert werden. In einer erfolgreichen Pilotstudie konnte durch die RIST Therapie in 38% der Patienten eine Langzeitremission erzielt werden. Um diesen vielversprechenden Ansatz weiter zu entwickeln, rekrutieren zurzeit zwei klinische Studien zur RIST Therapie mit Regensburg als Studienzentrale: rezidivierte und therapie-refraktäre Neuroblastome (RIST-rNB-2011) und Pineoblastome (RIST-rPB-2015-P).
Das Ziel dieses Forschungsprojektes ist es, die molekulare Grundlage der RIST Therapie zu studieren, um so die anti-tumorale Wirkung und Verträglichkeit weiter verbessern zu können. Weiterhin sollen molekulare Marker identifiziert werden, die vorhersagen, welche Patienten auf die Therapie ansprechen können.
(2) RNA-Biologie in pädiatrischen Tumorerkrankungen: RNA-bindende Proteine regulieren eine Vielzahl essentieller zellulärer Prozesse, wie zum Beispiel die Synthese von Proteinen oder die Entstehung nicht-kodierender RNA, die eine wichtige regulatorische Funktion bei der Genexpression ausüben. In den letzten Jahren wurde deutlich, dass fehlregulierte RNA-bindende Proteine zur Entstehung, Therapieresistenz und Metastasierung von Krebszellen beitragen.
Das Ziel dieses Forschungsprojektes ist es, die Funktion von RNA-bindenden Proteinen in der Krebszelle zu studieren, um neue therapeutisches Ansätze zu finden, die die Entstehung, Therapieresistenz und Metastasierung von Krebszellen hemmen.
Die Forschung der Arbeitsgruppe Pädiatrische und Molekulare Onkologie wird durch den Verein für krebskranke und behinderte Kinder e.V. (VKKK) unterstützt.
AG Poeck - Gezielte Immunmodulation in Krebs
Deutsch
Meine Arbeitsgruppe beschäftigt sich mit der Erforschung von Anti-Tumor-Antworten, der Auslösung von Resistenzmechanismen sowie der Geweberegeneration bei der Behandlung von Krebspatienten mittels (i) allogener Stammzelltransplantation, (ii) zellulären Therapien mit genetisch modifizierten T Zellen oder Makrophagen mit synthetischen, chimären Antigenrezeptoren (CAR) und (iii) Immun-Checkpoint-Inhibitoren (ICI). Im Speziellen möchten wir dabei ermitteln, welche Bedeutung spezifische Signalwege des angeborenen Immunsystems, die verschiedenen Komponenten des Mikrobioms und vom Tumor-abstammende Faktoren hinsichtlich Therapieerfolg und Resistenzentstehung sowie für die Geweberegeneration im Kontext o.g. Immuntherapien besitzen. Bei weiterhin großen inter-individuellen Unterschieden im Therapieansprechen ist es das Ziel unserer Forschungsarbeit, obige Information für die Entwicklung neuer, gezielter Kombinationstherapien zur Verbesserung der Ansprechraten dieser Immuntherapien zu nutzen. Gleichzeitig sollen unerwünschte Immun-vermittelte Nebenwirkungen wie die Spender gegen Empfängerreaktion (engl. graft-versus-host disease (GVHD), Cytokine Release Syndrome (CRS), oder Autoimmunphänomene nach allogener Stammzelltransplantation, CAR Therapie oder ICI minimiert werden.
Über den wissenschaftlichen Leiter der Arbeitsgruppe
Hendrik Poeck studierte Humanmedizin an der Ludwig-Maximilians-Universität München und erwarb dort seinen Doktortitel („summa cum laude“) in der damaligen Arbeitsgruppe von Prof. Gunther Hartmann in der Abteilung für Klinische Pharmakologie (2006, Leitung: Prof. Dr. Stefan Endres), wo er zum Einfluss von „plasmazytoiden dendritischen Zellen (pDC) und immunstimulatorischer DNA auf B-Zellen des humanen Immunsystems“ forschte. Danach arbeitete er als Postdoc in der Arbeitsgruppe von Prof. Hartmann (jetzt Universitätsklinikum Bonn) und Prof. Ruland (Klinikum Rechts der Isar, Technische Universität München (TUM)) an molekularen Mechanismen zur Auslösung von Entzündungsprozessen und Antitumorantworten. 2012 schloss Prof. Poeck seine Habilitation erfolgreich im Fach Innere Medizin ab. Als Stipendiat der Alexander von Humboldt Stiftung führte er 2013 seine Forschungslaufbahn am Memorial Sloan Kettering Cancer Center (MSKCC) in New York (USA) fort. Hier widmete er sich im Labor von Prof. Marcel van den Brink der Rolle des Mikrobioms und des angeborenen Immunsystems bei der Entstehung der graft-vs.-host disease (GVHD) und des graft-vs. leukemia (GVL) Effektes nach allogener Stammzelltransplantation. 2015 kehrte Prof. Poeck an die TUM zurück und gründete mit Unterstützung der European Hematology Association (EHA) eine Nachwuchsgruppe, welche Resistenzmechanismen bei Krebszellen sowie die Geweberegeneration bei immuntherapeutischen Ansätzen untersucht. Im Jahr 2017 schloss er seine Facharztausbildung für Hämatologie-Onkologie am Klinikum rechts der Isar (TUM) ab und wurde Oberarzt an der Klinik und Poliklinik für Innere Medizin III. Zum 01. Februar 2020 hat Herr Poeck den Ruf auf die W2-Professur für Innere Medizin mit Schwerpunkt Stammzelltransplantations- und Tumorimmunologie an der Universität Regensburg angenommen.
Key Awards
- Seit 2020: Scientific Committee Member for Stem Cell Transplantation and Cellular Therapies of the American Society of Hematology (ASH): Selection of potential topics and speakers for the ASH annual meeting, identify future Members of the Committee and ASH Honorific Awards
- 2018: Admission into the EMBO Young Investigator Programme (supports the best young researchers in the life sciences in Europe and beyond).
- 2017: Mechthild-Harf Research grant by DKMS
- 2016: European Hematology Association (EHA) Research Grant
- 2013: Feodor Lynen Research Fellowship for Experienced Researchers (Alexander von Humboldt-Foundation)
- 2010: Walther-Schulz Research Award
- 2009: Vincenz-Czerny Research Award by the German society for Hematology and Oncology (DGHO)
- 2006: Kulturpreis Bayern by Bavarian State Ministry of Sciences, Research and the Arts and E.ON Bayern AG (for the best doctoral thesis of the LMU)
- 2004: Harvard-Scholarship by the LMU-HMI-Alliance
- 2003-2005: Scholarship for gifted students of the Bayer Science and education Foundation
- 2003: Carl-Duisberg Scholarship
Ausgewählte Publikationen
- Poeck H, Wintges A, Dahl S, Bassermann F, Haas T, Heidegger S.. Tumor cell-intrinsic RIG-I signaling governs synergistic effects of immunogenic cancer therapies and checkpoint inhibitors. Eur J Immunol., 2021, in press, DOI: 10.1002/eji.202049158
- Heidegger S*#, Wintges A *, Stritzke F, Bek S, Steiger K, Koenig PA, Göttert S, Engleitner T, Öllinger R, Nedelko T, Fischer JC, Makarov V, Winter C, Rad R, van den Brink MRM, Ruland J, Bassermann F, Chan TA, Haas T*, Poeck H*#. RIG-I activation is critical for responsiveness to checkpoint blockade. Sci Immunol., 2019 Sep 13;4(39).
- Fischer JC, Bscheider M, Göttert S, Thiele Orberg E, Combs SE, Bassermann F, Heidegger S, Haas T, Poeck H. Type I interferon signaling before hematopoietic stem cell transplantation lowers donor T cell activation via reduced allogenicity of recipient cells. Sci Rep. 2019 Oct 18;9(1):14955.
- Bek S, Stritzke F, Wintges A, Nedelko T, Böhmer D.F.R, Fischer JC, Haas T, Poeck H*# & Simon Heidegger*#. Targeting intrinsic RIG-I signaling turns melanoma cells into type I interferon-releasing cellular antitumor vaccines. (*these authors contributed equally). Oncoimmunology [2162-4011] Bek, Sarah J.:2019 S.:1 -9.
- Heidegger S*#, Kreppel D*, Bscheider M, Stritzke F, Nedelko T, Wintges A, Bek S,Fischer JC, Graalman T, Kalinke U, Bassermann F, Haas T*, Poeck H*#. RIG-I activating immunostimulatory RNA boosts the efficacy of anticancer vaccines and synergizes with checkpoint blockade. EBioMedicine. 2019 Mar6. pii: S2352-3964(19)30135
- Fischer JC*, Otten V*, Drees C, Schmickl M, Heidegger S, Beyaert G, van Loo G, Li XC, Peschel C, Schmidt-Supprian M, Haas T, Spoerl S*#, Poeck H*#. NF-κB regulator A20 modulates thymic regulatory T cell development; J Immunol. 2017 Oct 1;199(7):2356-2365.
- Fischer JC*, M Bscheider*, Gabriel Eisenkolb*, Lin CC, Wintges A, Otten V, Lindemans CA, Heidegger S, Rudelius M, Monette S, Porosnicu Rodriguez K, Calafiore M, Liebermann S, Liu C, Lienenklaus S, Weiss S, Kalinke U, Ruland J, Peschel C, Shono Y, Docampo M, Velardi E, Jenq R, Hanash A, Dudakov JA, Haas T, van den Brink MR *# and Poeck H*#. RIG-I/MAVS and STING signaling promote epithelial integrity during irradiation and immune-medited tissue injury; Sci Transl Med. 2017 Apr 19;9(386).
- Fischer JC, Wintges A, Haas T, Poeck H. Assessment of mucosal integrity by quantifying neutrophil granulocyte influx in murine models of acute intestinal injury; Cell Immunol. 2017 Apr 11. pii: S0008-8749(17)30050-3
- Dann A*, Poeck H*, Maihofer C, Endres S, Kalinke U, Knobeloch KP, Akira S, Waisman A, Hartmann G and Prinz M. Cytosolic RIG-I-like helicases act as negative regulators of sterile inflammation in the CNS. Nat Neurosci 2011 Dec 4;15(1):98-106. doi: 10.1038/nn.2964 (*These authors contributed equally)
- Poeck H*, Bscheider M*, Gross O*, Roth S, Finger K, Hannesschläger N, Schlee M, Rebsamen M, Rothenfusser S, Barchet W, Akira S, Inoue S, Endres S, Peschel C, Hartmann G*, Hornung V* and Ruland J* . Recognition of RNA virus by RIG-I results in activation of CARD9 and inflammasome signaling for interleukin 1beta production. Nat Immunol. 2010 Jan;11(1):63-9. (*These authors contributed equally)
- Besch R*, Poeck H*, Hohenauer T, Senft D, Häcker G, Berking C, Hornung V, Endres S, Ruzicka T, Rothenfusser S, Hartmann G. Proapoptotic signalling by RIG-I and MDA-5 results in type I interferon independent apoptosis in melanoma. J Clin Invest. 2009, Aug; 119(8):2399-411*These authors contributed equally
- Gross O*, Poeck H*, Bscheider M, Dostert C, Hannesschläger N, Endres S, Hartmann G, Tardivel A, Schweighoffer E, Tybulewicz V, Mocsai A, Tschopp J, Ruland J. Syk kinase signalling couples to the Nlrp3 inflammasome for anti-fungal host defence. Nature. 2009 May 21;459(7245):433-6. (*These authors contributed equally)
- Poeck H*, Besch R*, Maihoefer C, Renn M, Tormo D, Morskaya S, Kirschnek S, Gaffal E, Landsberg J, Hellmuth J, Schmidt A, Anz D, Bscheider M, Schwerd T, Berking C, Bourquin C, Kalinke U, Kremmer E, Kato H, Akira S, Meyers R, Häcker G, Neuenhahn M, Busch D, Ruland J, Rothenfusser S, Prinz M, Hornung V, Endres S, Tüting T & Hartmann G. 5’-triphosphate siRNA: turning gene-silencing and RIG-I activation against melanoma. Nat Med. 2008 Nov;14
Research Funding of ongoing projects
- DFG (Collaborative Research Center (CRC 1371 (2019-2023, PI, P05 “Role of innate immune signals and microbiota on intestinal epithelial regeneration in graft-versus host disease”);
- DFG (Collaborative Research Center (CRC 1335 (2018-2022, PI, P14 “Mechanisms of immune evasion caused by aberrant innate nucleic acid receptor signaling in tumor cells and their tumor microenvironment);
- DFG (Collaborative Research Center (CRC TRR 221 (2020-2022, Associated member, PI “Targeting cGAS-STING in allo-HSCT”)
- DKMS “PROTECTOR” (2018-2021, PI)
- Melanoma Research Alliance (2019-2022, PI, “Microbial metabolites in immunotherapy of malignant melanoma”, joint Project with PD Dr. Simon Heidegger),
- EMBO Young Investigator Program (2018-2023, PI, “Role of cytosolic nucleic acid receptors in cancer immunotherapy”; travel-, meeting-, lab retreat-, sequencing, leadership and PhD course support)
- Else Kröner-Research College Regensburg – Interdisciplinary Translational Immuno-Oncology (2020-2023; PI, “Role of innate immune signaling in epithelial regeneration”)
- Bavarian State Ministry of Science and Art for the promotion of Corona research projects (PI, “Microbiome and COVID-19”, 2020-2021)
Regular hashtags that describe the group environment:
- International, friendly, open-minded, motivated, creative, diligent, hard-working, social.
Scientific keywords that describe the group and its research:
- Innate immunity, microbiome, cancer therapy resistance and response, immunotherapy, stem cell transplantation, genetic and synthetic engineering.
Kontakt
Prof. Dr. Hendrik Poeck | Universitätsklinikum Regensburg | Klinik und Poliklinik für Innere Medizin III (Hämatologie und Onkologie)
Franz-Josef-Strauß-Allee 11 | 93053 Regensburg
T: +49 941 944-5542 (Sekretariat) | F: +49 941 944-5543 | hendrik.poeck(at)ukr.de
English
The work in my laboratory is centered on identifying molecular mechanisms of cancer therapy resistance and success as well as regenerative responses during immunotherapeutic treatments including allogeneic hematopoietic stem cell transplantation (allo-HSCT), cellular therapies (chimeric antigen receptor (CAR) T cells or Macrophages), immune checkpoint blockade (ICB) and certain chemotherapies. Specifically, we aim to understand how pattern recognition receptors (PRRs) of the innate immune system can promote or inhibit these responses and to what extent this is impacted by environmental (e.g. microbiome) and cell-derived factors (e.g. extracellular vesicles). By gaining a deeper understanding of the modulation of anti-tumor and regenerative responses, my goal is to develop new combinatorial strategies to improve existing cancer immunotherapies to address the unsolved clinical problem of great inter-individual response variability while ameliorating unwanted immune-mediated inflammatory tissue damage (such as graft-versus host disease (GVHD), cytokine release syndrome (CRS) or ICB–mediated autoinflammation).
About the Principal Investigator
Hendrik Poeck studied Medicine at the Ludwig-Maximilians-Universität München and finished his doctoral thesis (2006, grade: “summa cum laude”) on the “the influence of plasmacytoid dendritic cells and immunostimulatory DNA on primary human B cells” in the former research group of Prof. Gunther Hartmann in the Division of Clinical Pharmacology (head: Prof. Dr. Stefan Endres). He then worked as a postdoc in the research groups of Prof. Hartmann (now University Hospital Bonn) and Prof. Jürgen Ruland (Klinikum Rechts der Isar, Technische Universität München (TUM)) which centered around molecular mechanisms of antitumor responses and inflammation. In 2012, he successfully completed his postdoctoral thesis (habilitation). In 2013, and as a scholar of the Alexander von Humboldt-Foundation, he worked as a visiting scientist in the laboratory of Prof. Marcel van den Brink at Memorial Sloan Kettering Cancer Center (MSKCC) in New York (USA). There, he focused on the role of innate immunity and the microbiome during graft-vs.-host disease (GVHD) and graft-vs. tumor (GVT) responses following allogeneic hematopoietic stem cell transplantation (allo-HSCT). In 2015, he moved back to TUM and - supported by a research grant of the European Hematology Association (EHA)- started his own research group, which focuses on molecular mechanisms of cancer therapy resistance and success as well as regenerative responses in the intestinal epithelium during immunotherapeutic treatment approaches. In 2017, he completed his clinical specialization in Internal Medicine, Hematology and Oncology at TUM and since 2018 works as senior physician in adult stem cell transplantation and immunotherapy. In February 2020, he became appointed as Associate Professor of Stem Cell Transplantation and Tumor Immunology at the University Hospital Regensburg.
Key Awards
- Since 2020: Scientific Committee Member for Stem Cell Transplantation and Cellular Therapies of the American Society of Hematology (ASH): Selection of potential topics and speakers for the ASH annual meeting, identify future Members of the Committee and ASH Honorific Awards
- 2018: Admission into the EMBO Young Investigator Programme (supports the best young researchers in the life sciences in Europe and beyond).
- 2017: Mechthild-Harf Research grant by DKMS
- 2016: European Hematology Association (EHA) Research Grant
- 2013: Feodor Lynen Research Fellowship for Experienced Researchers (Alexander von Humboldt-Foundation)
- 2010: Walther-Schulz Research Award
- 2009: Vincenz-Czerny Research Award by the German society for Hematology and Oncology (DGHO)
- 2006: Kulturpreis Bayern by Bavarian State Ministry of Sciences, Research and the Arts and E.ON Bayern AG (for the best doctoral thesis of the LMU)
- 2004: Harvard-Scholarship by the LMU-HMI-Alliance
- 2003-2005: Scholarship for gifted students of the Bayer Science and education Foundation
- 2003: Carl-Duisberg Scholarship
Selected publications
- Poeck H, Wintges A, Dahl S, Bassermann F, Haas T, Heidegger S.. Tumor cell-intrinsic RIG-I signaling governs synergistic effects of immunogenic cancer therapies and checkpoint inhibitors. Eur J Immunol., 2021, in press, DOI: 10.1002/eji.202049158
- Heidegger S*#, Wintges A *, Stritzke F, Bek S, Steiger K, Koenig PA, Göttert S, Engleitner T, Öllinger R, Nedelko T, Fischer JC, Makarov V, Winter C, Rad R, van den Brink MRM, Ruland J, Bassermann F, Chan TA, Haas T*, Poeck H*#. RIG-I activation is critical for responsiveness to checkpoint blockade. Sci Immunol., 2019 Sep 13;4(39).
- Fischer JC, Bscheider M, Göttert S, Thiele Orberg E, Combs SE, Bassermann F, Heidegger S, Haas T, Poeck H. Type I interferon signaling before hematopoietic stem cell transplantation lowers donor T cell activation via reduced allogenicity of recipient cells. Sci Rep. 2019 Oct 18;9(1):14955.
- Bek S, Stritzke F, Wintges A, Nedelko T, Böhmer D.F.R, Fischer JC, Haas T, Poeck H*# & Simon Heidegger*#. Targeting intrinsic RIG-I signaling turns melanoma cells into type I interferon-releasing cellular antitumor vaccines. (*these authors contributed equally). Oncoimmunology [2162-4011] Bek, Sarah J.:2019 S.:1 -9.
- Heidegger S*#, Kreppel D*, Bscheider M, Stritzke F, Nedelko T, Wintges A, Bek S,Fischer JC, Graalman T, Kalinke U, Bassermann F, Haas T*, Poeck H*#. RIG-I activating immunostimulatory RNA boosts the efficacy of anticancer vaccines and synergizes with checkpoint blockade. EBioMedicine. 2019 Mar6. pii: S2352-3964(19)30135
- Fischer JC*, Otten V*, Drees C, Schmickl M, Heidegger S, Beyaert G, van Loo G, Li XC, Peschel C, Schmidt-Supprian M, Haas T, Spoerl S*#, Poeck H*#. NF-κB regulator A20 modulates thymic regulatory T cell development; J Immunol. 2017 Oct 1;199(7):2356-2365.
- Fischer JC*, M Bscheider*, Gabriel Eisenkolb*, Lin CC, Wintges A, Otten V, Lindemans CA, Heidegger S, Rudelius M, Monette S, Porosnicu Rodriguez K, Calafiore M, Liebermann S, Liu C, Lienenklaus S, Weiss S, Kalinke U, Ruland J, Peschel C, Shono Y, Docampo M, Velardi E, Jenq R, Hanash A, Dudakov JA, Haas T, van den Brink MR *# and Poeck H*#. RIG-I/MAVS and STING signaling promote epithelial integrity during irradiation and immune-medited tissue injury; Sci Transl Med. 2017 Apr 19;9(386).
- Fischer JC, Wintges A, Haas T, Poeck H. Assessment of mucosal integrity by quantifying neutrophil granulocyte influx in murine models of acute intestinal injury; Cell Immunol. 2017 Apr 11. pii: S0008-8749(17)30050-3
- Dann A*, Poeck H*, Maihofer C, Endres S, Kalinke U, Knobeloch KP, Akira S, Waisman A, Hartmann G and Prinz M. Cytosolic RIG-I-like helicases act as negative regulators of sterile inflammation in the CNS. Nat Neurosci 2011 Dec 4;15(1):98-106. doi: 10.1038/nn.2964 (*These authors contributed equally)
- Poeck H*, Bscheider M*, Gross O*, Roth S, Finger K, Hannesschläger N, Schlee M, Rebsamen M, Rothenfusser S, Barchet W, Akira S, Inoue S, Endres S, Peschel C, Hartmann G*, Hornung V* and Ruland J* . Recognition of RNA virus by RIG-I results in activation of CARD9 and inflammasome signaling for interleukin 1beta production. Nat Immunol. 2010 Jan;11(1):63-9. (*These authors contributed equally)
- Besch R*, Poeck H*, Hohenauer T, Senft D, Häcker G, Berking C, Hornung V, Endres S, Ruzicka T, Rothenfusser S, Hartmann G. Proapoptotic signalling by RIG-I and MDA-5 results in type I interferon independent apoptosis in melanoma. J Clin Invest. 2009, Aug; 119(8):2399-411*These authors contributed equally
- Gross O*, Poeck H*, Bscheider M, Dostert C, Hannesschläger N, Endres S, Hartmann G, Tardivel A, Schweighoffer E, Tybulewicz V, Mocsai A, Tschopp J, Ruland J. Syk kinase signalling couples to the Nlrp3 inflammasome for anti-fungal host defence. Nature. 2009 May 21;459(7245):433-6. (*These authors contributed equally)
- Poeck H*, Besch R*, Maihoefer C, Renn M, Tormo D, Morskaya S, Kirschnek S, Gaffal E, Landsberg J, Hellmuth J, Schmidt A, Anz D, Bscheider M, Schwerd T, Berking C, Bourquin C, Kalinke U, Kremmer E, Kato H, Akira S, Meyers R, Häcker G, Neuenhahn M, Busch D, Ruland J, Rothenfusser S, Prinz M, Hornung V, Endres S, Tüting T & Hartmann G. 5’-triphosphate siRNA: turning gene-silencing and RIG-I activation against melanoma. Nat Med. 2008 Nov;14
Research Funding of ongoing projects
- DFG (Collaborative Research Center (CRC 1371 (2019-2023, PI, P05 “Role of innate immune signals and microbiota on intestinal epithelial regeneration in graft-versus host disease”);
- DFG (Collaborative Research Center (CRC 1335 (2018-2022, PI, P14 “Mechanisms of immune evasion caused by aberrant innate nucleic acid receptor signaling in tumor cells and their tumor microenvironment);
- DFG (Collaborative Research Center (CRC TRR 221 (2020-2022, Associated member, PI “Targeting cGAS-STING in allo-HSCT”)
- DKMS “PROTECTOR” (2018-2021, PI)
- Melanoma Research Alliance (2019-2022, PI, “Microbial metabolites in immunotherapy of malignant melanoma”, joint Project with PD Dr. Simon Heidegger),
- EMBO Young Investigator Program (2018-2023, PI, “Role of cytosolic nucleic acid receptors in cancer immunotherapy”; travel-, meeting-, lab retreat-, sequencing, leadership and PhD course support)
- Else Kröner-Research College Regensburg – Interdisciplinary Translational Immuno-Oncology (2020-2023; PI, “Role of innate immune signaling in epithelial regeneration”)
- Bavarian State Ministry of Science and Art for the promotion of Corona research projects (PI, “Microbiome and COVID-19”, 2020-2021)
Regular hashtags that describe the group environment:
- International, friendly, open-minded, motivated, creative, diligent, hard-working, social.
Scientific keywords that describe the group and its research:
- Innate immunity, microbiome, cancer therapy resistance and response, immunotherapy, stem cell transplantation, genetic and synthetic engineering.
Contact
Prof. Dr. Hendrik Poeck | University Hospital Regensburg | Internal Medicine III - Hematology and Medical Oncology
Franz-Josef-Strauß-Allee 11 | 93053 Regensburg
T: +49 941 944-5542 (Sekretariat) | F: +49 941 944-5543 | hendrik.poeck(at)ukr.de
AG Pukrop - Mechanismen der Organmetastasierung
Leitung der Arbeitsgruppe:
- Prof. Dr. Tobias Pukrop
Mitglieder der Arbeitsgruppe:
- Dr. rer. nat. Raquel Blazquez
- Dr. rer. nat. Daniela Sparrer
- Dr. med. Florian Lüke
- Laura Trigueros
- Hannah Endres
- Elena Vollmer
- Gunnar Müller
Tobias Pukrop gründete sein Labor in Regensburg im Jahr 2015. Bei seiner früheren Forschung in Göttingen fand er heraus, dass die Mikroumgebung von Tumoren (speziell im Fall von Makrophagen/Mikroglia und Astrozyten) Krebszellen während der Kolonisierung des ZNS unterstützt. Aktuell konzentriert sich unser Labor auf die Erforschung der Mechanismen, welche die Metastasierung in residenten Gliazellen fördern. Das zugrundeliegende Ziel ist die künftige Entwicklung effektiver Therapien, die die Kolonisierung des ZNS durch Krebszellen stoppen bzw. verhindern können. Seit kurzem interessieren wir uns auch für die Mechanismen, die Krebszellen anwenden um sich nach der Besiedlung von Zielorganen gegen phagozytischen Attacken zu wehren. Im Detail konzentrieren wir uns dabei auf die Rolle des Glutathion (GSH) Metabolismus bezüglich des Kolonisationspotentials verschiedener Tumorzelllinien. Unser neuestes Projekt befasst sich mit der Rolle von Zelladhesionsfaktoren und Zellpolaritätsproteinen als potentielle Ziele in durch epitheliale Tumore verursachten Hirnmetastasen.
Etablierte Methoden
Organotypisches 3D-Ko-Kultur ex vivo Modell
Der entscheidende Schritt bei der Metastasierung des ZNS scheint die Auseinandersetzung zwischen von anderen Organen stammenden Krebszellen mit der Mikroumgebung von Zielorganen zu sein. Immer mehr Daten zeigen, dass diese Konkurrenzsituation oftmals die Apoptose der prä-metastatischen Zellen zur Folge hat und somit eine erfolgreiche Metastasierung verhindert wird. Jedoch ist dieser scheinbar bedeutende Schritt bislang noch kaum untersucht. Daher haben wir ein neues experimentelles Modell entwickelt, um unsere Kenntnisse über diesen Schritt der Metastasierung zu erweitern. Dabei wird ein organotypischer Hirnschnitt zusammen mit einem 3D Tumorzell-Plug eingebettet in Matrigel für 3-4 Tage kultiviert. Dieses Modell simuliert speziell die Interaktion von Krebszellen und Gliazellen am Übergang von Hirnparenchym und metastatischem Gewebe. Somit erlaubt uns diese Ko-Kultur Methode die Visualisierung und/oder Manipulation der Interaktionen in dieser wichtigen Zone. Außerdem können wir Hirngewebe von genetisch modifizierten Mäusen bzw. Tumorzellen verwenden, um die Rolle von bestimmten Genen innerhalb der Mikroumgebung oder in Krebszellen genauer zu untersuchen. Ein weiterer Vorteil dieses Modells ist die Verringerung der Anzahl an benötigten Versuchstieren. Es ist besonders von Nutzen bei der Bestimmung der Invasivität verschiedener Tumorzelllinien in das Hirnparenchym sowie um die Auswirkungen spezifischer Behandlungen gegen das Ausbreiten von Hirnmetastasen während des letzten und kritischsten Schritts der Metastasierung zu untersuchen.
Stereotaktische intrakortikale Injektion
Unter einem syngenen Mausmodell wird die Transplantation von murinen Tumorzellen in Tiere mit dem gleichen genetischen Hintergrund verstanden. Diese Modelle stellen eine wirkungsvolle Methode dar um zu untersuchen, wie Tumorzellen in Anwesenheit eines funktionierenden Immunsystems metastasieren. In diesem Rahmen sind sie von hoher Bedeutung für Studien, die sich mit zielgerichteten immunologisch-basierten Therapien beschäftigen, entweder alleine oder in Kombination mit anderen Wirkstoffen, welche das Immunsystems modifizieren können um Krebszellen zu erkennen und zu zerstören. Um die finalen Schritte im Metastasierungsprozess zu untersuchen, haben wir ein in vivo Modell etabliert, bei dem Krebszellen stereotaktisch in die kortikale Region von syngenen Mäusen injiziert werden. Dieser Ansatz hat mehrere Vorteile: der experimentelle Aufbau ist reproduzierbar, er erlaubt die präklinische Untersuchung der Kolonisation und die Effekte von therapeutischen Ansätzen sind messbar.
Laufende Forschungsprojekte
- DFG Projekt im Rahmen der Forschergruppe (FOR 2127) „Selektion und Adaptation während der metastatischen Krebsprogression“ zusammen mit Prof. Christoph Klein zum Thema „Einfluss von Makrophage-/Mikroglia-IL4R-Aktivierung auf ZNS-Kolonisierung“.
- DFG Projekt zusammen mit Dr. Neta Erez (Tel Aviv University) zum Thema „Die funktionelle Rolle der Astrogliose und Neuro-Inflammation in Hirnmetastasen des Malignen Melanoms“.
- Sonderforschungsbereich Transregio 305 "Striking a moving target: From mechanisms of metastatic organ colonisation to novel systemic therapies"
Abgeschlossene Forschungsprojekte
- BMBF Verbundprojekt „e:Bio Metastasys“ über Hirnmetastasierung.
- DFG Projekt im Rahmen der FOR 942 zusammen mit Prof. Claudia Binder und Prof. Uwe K. Hanisch.
- Projekt im Rahmen des Württembergischen Krebspreises über Hirnmetastasierung.
AG Schmidl - Genregulation in Immunzellen
Leitung der Arbeitsgruppe:
- Dr. rer. nat. Christian Schmidl
Mitglieder der Arbeitsgruppe:
- Dania Riegel, M.Sc. (Doktorandin)
- Lukas Wöhrl, B.Sc. (Masterand)
- Manuela Kovacs-Sautter (MTA)
- Brigitte Wild (MTA)
T-Lymphozyten können Tumoren erkennen und bekämpfen, doch das suppressive Tumormikromilieu kann die Funktion von tumorinfiltrierenden T-Lymphozyten (TILs) einschränken. Epigenetische Mechanismen wie die Modifikation und Verpackung der DNA tragen dazu bei, zelluläre Programme zu etablieren und aufrecht zu erhalten. Es wird vermutet, dass ebendiese Mechanismen auch dazu beitragen TILs in ihrem inaktiven Zustand zu „fixieren“, und somit erfolgreiche Behandlungen mit Immuntherapeutika, welche das körpereigene Immunsystem zur Tumorbekämpfung stimulieren sollen, verhindern. Wir versuchen die molekularen Vorgänge, die zur Immunzellfehlfunktion führen besser zu verstehen, um Wege für neue immuntherapeutische Behandlungsmethoden zu finden. Derzeit hat die Entwicklung von Einzelzell-Sequenzierungsmethoden weitgehend unser Verständnis von Zell-zu-Zell-Variabilität, zelluläre Plastizität und Heterogenität entschieden erweitert. Wir sind daran interessiert, Einzelzellanalysen zu verwenden und weiter zu entwickeln, um heterogene und hochdynamische Zellpopulationen wie Immun- und Krebszellen zu untersuchen.
Etablierte Methoden
- Bulk- und Einzelzellanalysen der Chromatinzugänglichkeit
- Chromatin Immunopräzipitation im Kombination mit „next generation sequencing“ (ChIP-seq)
- Genomeditierungen in pimären Immunzellen mittels CRISPR
- Bioinformatische Analysen von Sequenzierdaten
Laufende Forschungsprojekte
-
RCI Nachwuchsgruppe mit dem Schwerpunkt „Genregulation in Immunzellen“
-
DFG Projekt „Epigenom-Prägung von tumorinfiltrierenden T-Zellen durch das Tumormilieu“
AG Spang - Computational Diagnostics Group
Leitung der Arbeitsgruppe:
- Prof. Dr. Rainer Spang
Mitglieder der Arbeitsgruppe:
- Dr. Michael Altenbuchinger
- Dr. Claudio Lottaz
- Christian Kohler
- Thorsten Rehberg
- Franziska Görtler
- Rudi Schill
- Michael Huttner
Rainer Spang gründete seine Computational Diagnostics Gruppe 2002 am MPI für Molekulare Genetik in Berlin. 2006 zog die Gruppe nach Regensburg um. Die Spang-Gruppe entwickelt statistische Modelle und prädiktive Verfahren des maschinellen Lernens zur Analyse von Tumorexpressionsprofilen (Transkriptom, Proteom, und Metabolom). Die entwickelten Tools erstrecken sich von molekularen Klassifikatoren, über Verfahren zur Schätzung der zellulären Zusammensetzung von Tumorgeweben (Digital Tissue Deconvolution), bis hin zu Modellen der Signaltransduktion in Tumoren. Onkologisch liegt unser Interessensschwerpunkt auf diffus großzelligen B-Zell- Lymphomen (DLBCL). Wir entwickelten Klassifikatoren zur Unterscheidung des DLBCL von anderen Lymphomen und zu dessen molekularen Subtypisierung. Rainer Spang ist Sprecher des BMBF-Verbundes „MMML-Demonstrator“ zur Erforschung der Systembiologie von Lymphomen. Unser neuestes Projekt befasst sich mit stochastischen Modellen der Progression von Tumorgenomen und Tumorepigenomen während der Metastasierung.
Etablierte Methoden
- Bioinformatikanalyse der Transkriptome, Proteome und Metabolome von Tumoren
- Analyse von Microarray, Nanostring und RNAseq Daten
- Molekulare Klassifikatoren zur Diagnose, Prognose und zur Vorhersage des Therapieansprechens
- Schätzung der zellulären Zusammensetzung von Geweben (Digital Tissue Deconvolution)
- Modelle der Signaltransduktion in Tumoren (Nested Effects Models)
- Modelle der parakrinen Tumor-Stroma-Kommunikation (Causal Effect Estimation)
Methoden in Entwicklung
- Tumorproressionsmodelle (Mutual Hazard Networks)
- Spatial Transcriptomics Tools
- Analyse von Einzelzellgenomen und Einzelzelltraskriptomen
- Schätzung zelltypspezifischer Expression in Geweben
Laufende Forschungsprojekte
- DFG Projekt im Rahmen der Forschergruppe (FOR 2127) „Selektion und Adaptation während der metastatischen Krebsprogression“ zusammen mit Prof. Michael Rehli zum Thema „Progression des Tumor Epigenoms“.
- BMBF Verbundprojekt „eMed MMML-Demonstrator“ über die Systembiologie von Lymphomen (Sprecher und PI von Projekten zu (a) prognostischen Signaturen beim DLBCL, (b) Kommunikation zwischen Lymphomzellen und tumorassozierten Makrophagen (zusammen mit Tobias Pukrop, Wolfram Gronwald, Katja Detmer-Wilde und Peter Oefner) und (c) zur Entwicklung der Data-Sharing-Plattform LYMMML
AG Tumor- und Immunzell-Metabolismus (Molekulare Onkologie)
Leitung der Projekte:
- Prof. Dr. Marina Kreutz
- PD Dr. Kathrin Renner-Sattler
- Dr. Katrin Singer
- Dr. Katrin Peter
- Dr. Peter Siska
- Dr. Annette Schnell
Die AG Tumor- und Immunzell-Metabolismus (Molekulare Onkologie) beschäftigt sich mit dem Metabolismus von Immunzellen und Tumorzellen.
Eines unserer Projekte widmet sich hierbei der Frage, inwieweit der Vitamin D3 Metabolismus die Differenzierung von Immunzellen verändert. Wir konnten zeigen, dass Immunzellen aus dem Vorläufer 25-Hydroxyvitamin D3 den physiologisch aktiven Vitamin D3 Metaboliten 1,25-Dihydroxyvitamin D3 produzieren können. Dies induziert tolerogene dendritische Zellen und führt zur Differenzierung induzierter regulatorischer T Zellen. Dadurch könnten Vitamin D3 Metabolite in Entzündungssituationen als wichtiger Inhibitor der Immunreaktion wirken. Dies testen wir aktuell in einem Tiermodell der Knochenmarkstransplantation.
Ein weiteres Projekt beschäftigt sich mit der Interaktion von Immunzellen und Tumorzellen im Tumormilieu. Ein typisches Charakteristikum von Tumorzellen ist die gesteigerte Aufnahme und Abbau von Glukose („Warburg Effekt“), der zu einer Ansäuerung der Tumorumgebung und zu einer Akkumulation von Laktat führt. Wir und andere konnten zeigen, dass Laktat die Aktivierung von T-Zellen inhibiert. Neben der Analyse der metabolischen Interaktion von Tumor-und Immunzelle, interessiert uns besonders inwieweit T-Zellen durch genetische oder pharmakologische Modulation fit für den Einsatz im Tumor gemacht werden können. In aktuellen Arbeiten konnten wir zeigen, dass über pharmakologische Intervention die Laktatsekretion inhibiert und dadurch eine Immuntherapie mit Checkpoint-Inhibitoren verbessert werden kann. Die gezielte pharmakologische Inhibition der Glykolyse und/oder anderer Stoffwechselwege stellt somit eine therapeutische Möglichkeit dar, die Eliminierung des Tumors direkt und/oder durch eine Aktivierung des Immunsystems zu erreichen.
AG Tumorvakzinierung mittels onkolytischer Viren
Leitung der Arbeitsgruppe:
- Prof. Dr. Barbara Schmidt
Mitglieder der Arbeitsgruppe:
- Dr. rer. nat. Philipp Schuster
- Anette Rohrhofer
Die Arbeitsgruppe von Prof. Barbara Schmidt beschäftigt sich am Institut für Mikrobiologie und Hygiene der Universität Regensburg mit der Infektionsimmunologie von Herpes simplex Virus Typ 1 (HSV-1) und Humanem Immundefizienzvirus Typ 1 (HIV-1). Neben der klinischen Virologie stehen insbesondere aktuelle Fragestellungen zu onkolytischen Viren im Mittelpunkt des Interesses.
Onkolytische Viren sind Viren, die Tumorzellen infizieren und diese dadurch zerstören. Ende 2015 wurde das erste onkolytische Herpesvirus (T-VEC) für die Therapie von metastasiertem Malignem Melanom zugelassen; weitere onkolytische Viren sind in der Testung.
Die Arbeitsgruppe von Prof. Barbara Schmidt untersucht die onkolytische Aktivität von Herpesviren anhand von Melanomzelllinien. Diese Grundlagenforschung wird in Kooperation mit externen und internen Arbeitsgruppen durchgeführt.
Ziel der Arbeitsgruppe ist es, die Mechanismen der virusbedingten Onkolyse zu erforschen. Weiterhin soll ein onkolytisches Herpesvirus so weiterentwickelt werden, dass es Tumorantigene exprimiert und dadurch die adaptive Immunabwehr insbesondere von CD8+ T-Zellen effizienter als bisher adressiert. Es geht also um die Entwicklung einer onkolytischen Melanomvakzine.
Externe Kooperationspartner sind die Harvard Medical School und das Department für Dermatologie der Friedrich-Alexander-Universität Erlangen-Nürnberg. Interne Kooperationen bestehen mit Dr. rer. nat. Melanie Werner-Klein, dem Regensburger Centrum für Interventionelle Immunologie (RCI) und Institut für Immunologie der Universität Regensburg, und PD Dr. Sebastian Haferkamp, Klinik und Poliklinik für Dermatologie, Universitätsklinikum Regensburg.
www.imhr.de/forschung/arbeitsgruppen/prof-dr-barbara-schmidt
AG Thomas - T-Zell-Immuntherapie
Leitung der Projekte:
- Priv.-Doz. Dr. med. Simone Thomas
- Dr. Sebastian Klobuch
Die AG T-Zell-Immuntherapie beschäftigt sich mit der Entwicklung von T-Zell-Immuntherapien im Kontext der allogenen Stammzelltransplantation.
Der Fokus liegt auf der Generierung von Leukämie-spezifischen T-Lymphozyten mittels T-Zell-Rezeptoren, welche wir aus Leukämie-spezifischen T-Zellen isolieren und in T-Zellen gesunder Spender transferieren. Als Zielstruktur für eine Leukämie-spezifische Immuntherapie konzentrieren wir uns auf allogene HLA-DP Antigene, welche zu den HLA-Klasse II Antigenen gehören. Im Gegensatz zu HLA-Klasse I und HLA-DQ und -DR Antigenen unterscheiden sich HLA-DP Antigene in einem hohen Prozentsatz (bis zu 80%) zwischen Stammzellspender und Patient, weshalb sie als Zielstruktur für eine T-Zell-Therapie genutzt werden können.
Ein weiteres Ziel der AG ist die Entwicklung von Mechanismen, die die Sicherheit einer T-Zell-Rezeptor basierten Immuntherapie nach allogener Stammzelltransplantation verbessern sollen. Im Detail konzentrieren wir uns dabei auf die Entwicklung von Mechanismen, die das gezielte Ein- und Ausschalten der T-Zell-Funktion erlauben.
Die AG T-Zell-Immuntherapie wird u.a. im Rahmen des Sonderforschungsbereichs Transregio 221 gefördert und gehört als klinische Kooperationsgruppe dem RCI Regensburger Centrum für Interventionelle Immunologie an.
Arbeitsgruppen LEX & ITEM-R
Leitung des LEX Lehrstuhls für Experimentelle Medizin und Toxikologie:
- Prof. Dr. Christoph Klein
Arbeitsgruppen des LEX Lehrstuhls für Experimentelle Medizin und Toxikologie:
- Prof. Dr. Christoph Klein - Evolution der systemischen Krebserkrankung
- Dr. Melanie Werner-Klein – Immunbiologie der frühen systemischen Krebserkrankung
- Dr. Hedayatollah Hosseini – Signalwege der Metastasierung
- Dr. Zbigniew Czyz – Einzelzell-Technologie
Leitung des ITEM-R Bereich Personalisierte Tumortherapie des Fraunhofer Institut für Experimentelle Medizin und Toxikologie Regensburg:
- Prof. Dr. Christoph Klein
Arbeitsgruppen des ITEM-R Bereich Personalisierte Tumortherapie des Fraunhofer Institut für Experimentelle Medizin und Toxikologie Regensburg:
- Dr. Bernhard Polzer – Zelluläre und molekulare Diagnostik
- Dr. Stefan Kirsch - Innovative molekulare Technologie und Biomarkeridentifizierung
- Dr. Christian Werno - Präklinische Therapiemodelle
- Dr. Kamran Honarnejad - Hochdurchsatz-Drug- und -Target-Discovery
- Dr. Jens Warfsmann - Bioinformatik und Datenmanagement
- Dr. Martin Hoffmann – Mathematische Modellierung von Krankheitsprozessen
www.item.fraunhofer.de/tumortherapie
Unser Ziel ist es, Patientinnen und Patienten vor der Entwicklung einer zu großen Metastasenlast und damit vor einem tödlichen Verlauf der Krankheit zu schützen. Hierfür erforschen wir einerseits in unseren vier multidisziplinären Lehrstuhl-Arbeitsgruppen die grundlegenden Mechanismen der systemischen Ausbreitung der Krebserkrankung und der Metastasenbildung. Wir wollen ein möglichst genaues Verständnis der Evolution einer Krebserkrankung erlangen, eine Stadien-spezifische Diagnostik entwickeln und die Krankheit auf molekularer Ebene verfolgen. Wir wollen lernen, für jedes Krankheitsstadium und jede individuelle Erkrankung die bestmögliche Therapie vorherzusagen und schließlich neue Therapien zu entwickeln. Zum anderen wollen auch für Patientinnen und Patienten in fortgeschritten Stadien lernen, wie ein weiteres Voranschreiten verhindert werden kann.
Um dies zu erreichen, entwickeln in wir Einzelzelltechnologien und zelluläre Modelle zur Untersuchung von disseminierten Krebszellen (DCCs), die im Körper des Patienten verstreut sind. Diese Technologien helfen uns, die molekularen Eigenschaften der DCCs zu identifizieren und zu charakterisieren, die es den DCCs erlauben, Monate bis Jahre oder gar Jahrzehnte nach der Erstdiagnose tödliche Metastasen zu bilden. Darüber hinaus etablieren wir in-vitro- und in-vivo-Modelle für die funktionelle Analyse von DCCs unter Berücksichtigung der Signalwege und der Immunbiologie der frühen systemischen Krebserkrankung. Um unsere Erkenntnisse in diagnostische oder therapeutische Anwendungen umzusetzen, betreiben wir ein diagnostisches Labor (systemic cancer progression laboratory, SCP-lab) zum Nachweis von systemisch gestreuten und zirkulierenden Tumorzellen und arbeiten mit dem lokalen Fraunhofer-Bereich für Personalisierte Tumortherapie (ITEM-Regensburg) zusammen. Dort werden die Erkenntnisse aus der Grundlagenforschung für die Anwendung in der Klinik im Bereich Diagnostik und Therapie weiterentwickelt. All diese Projekte verfolgen wir in enger Zusammenarbeit mit Kliniken am UKR, im Krankenhaus St. Josef und im Krankenhaus der Barmherzigen Brüder.
OTH Regensburg: Labor Technikfolgen und angewandte Ethik (LaTE)
Leitung des Labors:
- Prof. Dr. Karsten Weber
Mitglieder des Labors:
- Uta Bittner
- Vasilija Rolfes
- Ulrike Scorna
- Arne Sonar
- Peter Wegenschimmel
Krankheit und Gesundheit sind nicht allein medizinische, sondern ebenso soziale und ethische Kategorien. Technik wiederum verändert die Behandlung von Krankheiten und den Umgang mit Krankheit und Gesundheit. Das alles gilt insbesondere für Tumorerkrankungen, die nach wie vor zu den häufigsten Sterbeursachen gehören und allein dadurch besondere gesellschaftliche Aufmerksamkeit erzeugen. Die teilweise sehr kostenintensiven Behandlungen stellen ebenso wie Therapiefolgen, Sinnhaftigkeit von Therapien und Therapieabbrüchen schwerwiegende moralische Fragen. Neue KI-gestützte diagnostische Verfahren werfen Fragen der Verantwortung, des Professionsverständnisses und sogar erkenntnistheoretische Fragen auf.
LaTE wurde 2018 gegründet und ist ein Labor der Fakultät Allgemeinwissenschaften und Mikrosystemtechnik der Ostbayerischen Technischen Hochschule Regensburg (OTH Regensburg). Darüber hinaus ist es Gründungslabor des Regensburg Center of Health Sciences and Technology (RCHST, www.rchst.de) und Teil des Instituts für Sozialforschung und Technikfolgenabschätzung (IST, www.oth-regensburg.de/ist) der OTH Regensburg.
Methoden
- Ethische Evaluation mit MEESTAR oder Action Sheets
- Quantitative und qualitative Methoden der Sozialforschung
- Szenarienbildung
Laufende Forschungsprojekte
- Stakeholderperspektiven auf KI-unterstützte medizinische Entscheidungsfindung und Entwicklung ethischer Leitlinien für den Einsatz von KI-Systemen in der Medizin (KI & Ethik, in Kooperation mit einem KI-Bildanalyse Projekt im Labor ReMIC, geleitet von Prof. Dr. Palm).
www.hs-regensburg.de/labor-technikfolgenabschaetzung-angewandte-ethik
OTH Regensburg: Labor Regensburg Medical Image Computing (ReMIC)
Leitung des Labors:
- Prof. Dr. Christoph Palm
Mitglieder des Labors:
- Moritz Jungtäubl
- Johannes Maier
- Robert Mendel
- David Rauber
- Maximilian Weiherer
- Sümeyye R. Yildiran
- Martin Zorn
Das ReMIC entwickelt Methoden der Medizinischen Bildverarbeitung insbesondere zur computer-unterstützten Diagnostik. Dabei werden medizinische Bilddaten analysiert, um sie automatisch in bestimmte Klassen (z.B. Tumor/nicht-Tumor) oder um bestimmte Gewebearten (z.B. Tumore) zu segmentieren. Dabei kommen verstärkt Algorithmen aus dem Bereich der Künstlichen Intelligenz, insbesondere Deep Learning mit Künstlichen Neuronalen Netzen, zum Einsatz. Als Bilddaten kommen zweidimensionale Daten z.B. aus der Endoskopie genauso in Frage wie dreidimensionale CT- oder MRT-Volumina.
Das ReMIC wurde 2011 gegründet und ist ein Labor der Fakultät Informatik und Mathematik der Ostbayerischen Technischen Hochschule Regensburg (OTH Regensburg). Darüber hinaus ist es Gründungslabor des Regensburg Center of Biomedical Engineering (RCBE, www.rcbe.de) und des Regensburg Center of Health Sciences and Technology (RCHST, www.rchst.de).
Methoden
- Künstliche Neurale Netze, insbes. Deep Learning
- Computer-unterstützte Diagnose mit Hilfe von Maschinellem Lernen
- Segmentierung: Objekt Detektion und Delination
- Bildklassifikation und Merkmalsextraktion
- Bildregistrierung, linear und nicht-linear
- Virtuelle Realität, insbes. Haptische Komponenten
Laufende Forschungsprojekte
- Künstliche Intelligenz zur Auswertung medizinischer Bilddaten am Beispiel Früherkennung von Speiseröhrenkrebs (KI & Ethik, in Kooperation mit dem Labor Technikfolgen und angewandte Ethik, geleitet von Prof. Dr. Karsten Weber)